mle
최대가능도 추정값
설명
phat = mle(data,Name,Value)
예를 들어, 이름-값 인수 Distribution, pdf, logpdf 또는 nloglf 중 하나를 사용하여 분포 유형을 지정할 수 있습니다.
내장 분포에 대한 MLE를 계산하려면
Distribution을 사용하여 분포 유형을 지정하십시오. 예를 들어,'Distribution','Beta'사용자 지정 분포에 대한 MLE를 계산하려면
pdf,logpdf또는nloglf를 사용하여 분포를 정의하고Start를 사용하여 초기 모수 값을 지정하십시오.
예제
Distribution 이름-값 인수를 사용하여 지정한 내장 분포에 대해 MLE를 구합니다.
표본 데이터를 불러옵니다.
load carbig변수 MPG는 다양한 자동차 모델에 대한 갤런당 마일 주행거리를 포함합니다.
MPG 데이터에 대한 히스토그램을 그립니다.
histogram(MPG)

이 분포는 오른쪽 꼬리가 다소 깁니다. 정규분포와 같은 대칭 분포는 양호한 피팅이 아닐 수 있습니다.
MPG 데이터에 대한 버 제12종 분포의 모수를 추정합니다.
phat = mle(MPG,'Distribution','burr')
phat = 1×3
34.6447 3.7898 3.5722
스케일 모수 α에 대한 MLE는 34.6447입니다. 버 제12종 분포에 대한 두 개의 형태 모수 및 의 추정값은 각각 3.7898 및 3.5722입니다.
시행 횟수 = 20, 성공 확률 = 0.75를 사용하여 이항분포에서 100개의 임의 관측값을 생성합니다.
rng('default') % For reproducibility data = binornd(20,0.75,100,1);
시뮬레이션된 표본 데이터를 사용하여 성공 확률과 95% 신뢰한계를 추정합니다. 이항분포에 사용할 시행 횟수(NTrials)를 지정해야 합니다.
[phat,pci] = mle(data,'Distribution','binomial','NTrials',20, ... 'Alpha',.01)
phat = 0.7615
pci = 2×1
0.7361
0.7856
성공 확률에 대한 추정값은 0.7615이고 99% 신뢰구간의 하한 및 상한은 각각 0.7361 및 0.7856입니다. 이 구간은 데이터를 시뮬레이션하는 데 사용된 실제 값을 포함합니다.
자유도를 8로 하고 비중심성 모수를 3으로 하여 비중심 카이제곱 분포에서 크기가 1000인 표본 데이터를 생성합니다.
rng default % for reproducibility x = ncx2rnd(8,3,1000,1);
표본 데이터에서 비중심 카이제곱 분포에 대한 모수를 추정합니다. Distribution 이름-값 인수는 비중심 카이제곱 분포를 지원하지 않습니다. 따라서, pdf 이름-값 인수 및 ncx2pdf 함수를 사용하여 사용자 지정 비중심 카이제곱 pdf를 정의해야 합니다. 사용자 지정 분포에 대한 초기 모수 값(Start 이름-값 인수)도 지정해야 합니다.
[phat,pci] = mle(x,'pdf',@(x,v,d)ncx2pdf(x,v,d),'Start',[1,1])
phat = 1×2
8.1052 2.6693
pci = 2×2
7.1120 1.6025
9.0983 3.7362
자유도에 대한 추정값은 8.1052이고 비중심성 모수에 대한 추정값은 2.6693입니다. 자유도에 대한 95% 신뢰구간은 (7.1120,9.0983)이고 비중심성 모수에 대한 95% 신뢰구간은 (1.6025,3.7362)입니다. 신뢰구간은 각각 실제 모수 값인 8과 3을 포함합니다.
표본 데이터를 불러옵니다.
load('readmissiontimes.mat');데이터는 100명의 환자에 대한 재입원 횟수를 갖는 ReadmissionTime을 포함합니다. 이 데이터는 시뮬레이션된 데이터입니다.
스케일 모수 lambda와 형태 모수 k를 갖는 베이불 분포에 대해 사용자 지정 로그 pdf를 정의합니다.
custlogpdf = @(data,lambda,k) ...
log(k) - k*log(lambda) + (k-1)*log(data) - (data/lambda).^k;사용자 지정 분포의 모수를 추정하고 그 초기 모수 값(Start 이름-값 인수)을 지정합니다.
phat = mle(ReadmissionTime,'logpdf',custlogpdf,'Start',[1,0.75])
phat = 1×2
7.5727 1.4540
사용자 지정 분포의 스케일 모수와 형태 모수는 각각 7.5727 및 1.4540입니다.
표본 데이터를 불러옵니다.
load('readmissiontimes.mat')데이터는 100명의 환자에 대한 재입원 횟수를 갖는 ReadmissionTime을 포함합니다. 이 데이터는 시뮬레이션된 데이터입니다.
모수 lambda를 갖는 푸아송 분포에 대해 사용자 지정 음의 로그 가능도 함수를 정의합니다. 이때 1/lambda은 분포의 평균입니다. 사용자 지정 함수에 이러한 값을 사용하지 않는 경우에도, 함수가 중도절단 정보로 구성된 논리형 벡터와 데이터 도수로 구성된 정수 벡터를 받도록 정의해야 합니다.
custnloglf = @(lambda,data,cens,freq) ... - length(data)*log(lambda) + sum(lambda*data,'omitnan');
사용자 지정 분포의 모수를 추정하고 그 초기 모수 값(Start 이름-값 인수)을 지정합니다.
phat = mle(ReadmissionTime,'nloglf',custnloglf,'Start',0.05)
phat = 0.1462
자유도를 10을 하고 비중심성 모수를 5로 하여 비중심 카이제곱 분포에서 크기가 1000인 표본 데이터를 생성합니다.
rng('default') % For reproducibility x = ncx2rnd(10,5,1000,1);
비중심성 모수가 값 5에 고정되었다고 가정합니다. 표본 데이터에서 비중심 카이제곱 분포의 자유도를 추정합니다. 이를 수행하려면 pdf 이름-값 인수를 사용하여 사용자 지정 비중심 카이제곱 pdf를 정의해야 합니다.
[phat,pci] = mle(x,'pdf',@(x,v)ncx2pdf(x,v,5),'Start',1)
phat = 9.9307
pci = 2×1
9.5626
10.2989
비중심성 모수에 대한 추정값은 9.9307이고 95% 신뢰구간의 하한 및 상한은 각각 9.5626 및 10.2989입니다. 신뢰구간은 실제 모수 값인 10을 포함합니다.
스케일 모수를 카이제곱 분포에 추가하여 데이터 스케일에 맞게 조정하고 분포를 피팅합니다.
자유도를 5로 하여 카이제곱 분포에서 크기가 1000인 표본 데이터를 생성하고 인자를 100으로 하여 데이터를 스케일링합니다.
rng default % For reproducibility x = 100*chi2rnd(5,1000,1);
자유도와 스케일링 인자를 추정합니다. 이를 수행하려면 pdf 이름-값 인수를 사용하여 사용자 지정 카이제곱 확률 밀도 함수를 정의해야 합니다. 밀도 함수를 사용하려면 로 스케일링된 데이터에 대한 인자가 필요합니다.
[phat,pci] = mle(x,'pdf',@(x,v,s)chi2pdf(x/s,v)/s,'Start',[1,200])
phat = 1×2
5.1079 99.1681
pci = 2×2
4.6862 90.1215
5.5297 108.2146
자유도에 대한 추정값은 5.1079이고 스케일에 대한 추정값은 99.1681입니다. 자유도에 대한 95% 신뢰구간은 (4.6862,5.5279)이고 스케일 모수에 대한 95% 신뢰구간은 (90.1215,108.2146)입니다. 신뢰구간은 각각 실제 모수 값인 5와 100을 포함합니다.
표본 데이터를 불러옵니다.
load('readmissiontimes.mat');데이터는 100명의 환자에 대한 재입원 횟수를 갖는 ReadmissionTime을 포함합니다. 열 벡터 Censored는 각 환자에 대한 중도절단 정보를 포함합니다. 여기서 1은 우측 중도절단된 관측값을 나타내고, 0은 정확한 재입원 횟수가 관측되었음을 나타냅니다. 이 데이터는 시뮬레이션된 데이터입니다.
모수 lambda를 갖는 지수 분포에 대해 사용자 지정 확률 밀도 함수(pdf) 및 누적 분포 함수(cdf)를 정의합니다. 이때 1/lambda은 분포의 평균입니다. 분포를 중도절단된 데이터 세트에 피팅하려면 pdf 및 cdf를 모두 mle 함수에 전달해야 합니다.
custpdf = @(data,lambda) lambda*exp(-lambda*data); custcdf = @(data,lambda) 1-exp(-lambda*data);
중도절단된 표본 데이터에 대한 사용자 지정 분포의 모수 lambda를 추정합니다. 사용자 지정 분포에 대한 초기 모수 값(Start 이름-값 인수)을 지정합니다.
phat = mle(ReadmissionTime,'pdf',custpdf,'cdf',custcdf, ... 'Start',0.05,'Censoring',Censored)
phat = 0.1096
양측 중도절단 생존 데이터를 생성하고 데이터의 내장 분포에 대한 MLE를 구합니다. 그런 다음 MLE를 사용하여 확률 분포 객체를 만듭니다.
번바움-손더스(Birnbaum-Saunders) 분포에서 고장 시간을 생성합니다.
rng('default') % For reproducibility failuretime = random('BirnbaumSaunders',0.3,1,[100,1]);
연구가 시간 0.1에서 시작하고 시간 0.9에서 끝난다고 가정합니다. 이 가정은 0.1보다 작은 고장 시간은 좌측 중도절단되고 0.9보다 큰 고장 시간은 우측 중도절단됨을 의미합니다.
각 요소가 failuretime의 대응되는 관측값에 대한 중도절단 상태를 나타내는 벡터를 생성합니다. –1, 1 및 0을 사용하여 각각 좌측 중도절단된 관측값, 우측 중도절단된 관측값 및 완전히 관측된 관측값을 나타냅니다.
L = 0.1; U = 0.9; left_censored = (failuretime<L); right_censored = (failuretime>U); c = right_censored - left_censored;
양측 중도절단된 데이터에 대한 MLE를 구합니다. Censoring 이름-값 인수를 사용하여 중도절단 정보를 지정합니다.
phat = mle(failuretime,'Distribution','BirnbaumSaunders','Censoring',c)
phat = 1×2
0.2632 1.3040
makedist 함수를 통해 MLE를 사용하여 확률 분포 객체를 만듭니다.
pd = makedist('BirnbaumSaunders','beta',phat(1),'gamma',phat(2))
pd =
BirnbaumSaundersDistribution
Birnbaum-Saunders distribution
beta = 0.263184
gamma = 1.304
pd는 BirnbaumSaundersDistribution 객체입니다. pd의 객체 함수를 사용하여 분포를 실행하고 난수를 생성할 수 있습니다. 지원되는 객체 함수를 표시합니다.
methods(pd)
Methods for class prob.BirnbaumSaundersDistribution: cdf gather icdf iqr mean median negloglik paramci pdf plot proflik random std truncate var
예를 들어 mean 함수 및 var 함수를 사용하여 각각 분포의 평균 및 분산을 계산합니다.
mean(pd)
ans = 0.4869
var(pd)
ans = 0.3681
베이불 분포에 따라 기계의 고장 시간을 나타내는 표본 데이터를 생성합니다.
rng('default') % For reproducibility failureTimes = wblrnd(5,2,[200,1]);
관측된 고장 시간을 가장 가까운 초로 반올림한 값이 되도록 지정합니다.
observed = round(failureTimes);
observed는 구간 중도절단된 데이터입니다. observed의 관측값 t는 이벤트가 시간 t–0.5 이후 및 시간 t+0.5 이전에 발생했음을 나타냅니다.
중도절단 정보를 포함하는 2열 행렬을 생성합니다.
intervalTimes = [observed-0.5 observed+0.5];
고장 시간은 양수여야 합니다. eps보다 작은 값을 찾아서 eps로 변경합니다.
intervalTimes(intervalTimes < eps) = eps;
intervalTimes를 사용하여 베이불 분포 모수에 대한 MLE를 구합니다.
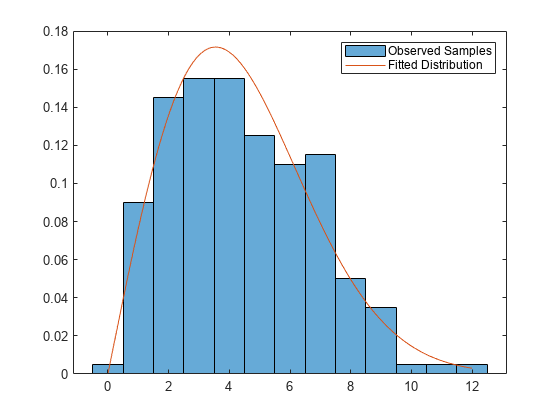
params = mle(intervalTimes,'Distribution','Weibull')
params = 1×2
5.0067 2.0049
결과를 플로팅합니다.
figure histogram(observed,'Normalization','pdf') hold on x = linspace(0,max(observed)); plot(x,wblpdf(x,params(1),params(2))) legend('Observed Samples','Fitted Distribution') hold off
유한 지지 범위를 가진 분포에서 표본을 생성하고, 반복 추정 과정에 대해 사용자 지정된 옵션을 사용하여 MLE를 구합니다.
확률 밀도가 0인 영역이 있는 분포의 경우 mle는 일부 모수에서 밀도가 0인 모수를 시도할 수 있으며 이로 인해 함수가 MLE를 구하지 못할 수 있습니다. 이 문제를 방지하기 위해, 유효하지 않은 함수 값을 검사하는 옵션을 끄고, mle 함수를 호출할 때 모수 한계를 지정할 수 있습니다.
스케일 모수를 1로 하고 형태 모수를 1로 하여 베이불 분포에서 크기가 1000인 표본 데이터를 생성합니다. 10을 더해 표본을 이동합니다.
rng('default') % For reproducibility data = wblrnd(1,1,[1000,1]) + 10; histogram(data,'Normalization','pdf')

히스토그램을 통해 10보다 작은 표본이 없음을 알 수 있습니다. 즉 분포는 10보다 작은 영역에서 확률 0을 가집니다. 이 분포는 위치에 대한 세 번째 모수를 포함하는 3-모수 베이불 분포입니다(Three-Parameter Weibull Distribution 참조).
3-모수 베이불 분포에 대한 확률 밀도 함수(pdf)를 정의합니다.
custompdf = @(x,a,b,c) wblpdf(x-c,a,b);
mle 함수를 사용하여 MLE를 구합니다. Options 이름-값 인수를 지정하여, 유효하지 않은 함수 값을 검사하는 옵션을 끕니다. 또한 LowerBound 및 UpperBound 이름-값 인수를 사용하여 모수 한계를 지정합니다. 스케일 모수와 형태 모수는 양수여야 하고, 위치 모수는 표본 데이터의 최솟값보다 작아야 합니다.
params = mle(data,'pdf',custompdf,'Start',[5 5 5], ... 'Options',statset('FunValCheck','off'), ... 'LowerBound',[0 0 -Inf],'UpperBound',[Inf Inf min(data)])
params = 1×3
1.0258 1.0618 10.0004
mle 함수는 3개 모수에 대한 정확한 추정값을 구합니다. 반복 과정에서의 사용자 지정 옵션 지정에 대한 자세한 내용은 예제 Three-Parameter Weibull Distribution 항목을 참조하십시오.
입력 인수
표본 데이터 및 중도절단 정보로, 표본 데이터로 구성된 벡터나 표본 데이터 및 중도절단 정보로 구성된 2열 행렬로 지정됩니다.
data 인수 또는 Censoring 이름-값 인수를 사용하여 표본 데이터에 대한 중도절단 정보를 지정할 수 있습니다. data가 2열 행렬인 경우 mle는 Censoring 인수 값을 무시합니다.
data의 관측값의 중도절단 유형에 따라 data를 벡터 또는 2열 행렬로 지정하십시오.
완전히 관측된 데이터 —
data를 표본 데이터로 구성된 벡터로 지정합니다.완전히 관측된 관측값, 좌측 중도절단된 관측값 또는 우측 중도절단된 관측값이 포함된 데이터 —
data를 표본 데이터로 구성된 벡터로 지정하고Censoring이름-값 인수를 각 관측값에 대한 중도절단 정보를 포함하는 벡터로 지정합니다.Censoring벡터는 각각 완전히 관측된 관측값, 좌측 중도절단된 관측값 및 우측 중도절단된 관측값을 나타내는 0, –1 및 1을 포함할 수 있습니다.구간 중도절단된 관측값을 포함하는 데이터 —
data를 표본 데이터 및 중도절단 정보로 구성된 2열 행렬로 지정합니다.data의 각 행은 각 관측값의 가능한 생존 시간 또는 고장 시간의 범위를 지정하며 다음 값 중 하나를 가질 수 있습니다.[t,t]—t에서 완전히 관측됨[–Inf,t]—t에서 좌측 중도절단됨[t,Inf]—t에서 우측 중도절단됨[t1,t2]—[t1,t2]에서 구간 중도절단됨. 여기서t1<t2임
중도절단된 관측값을 지원하는 내장 분포 목록은
Censoring항목을 참조하십시오.mle는data에 포함된NaN값을 무시합니다. 또한, 중도절단 벡터(Censoring)나 도수 벡터(Frequency)에NaN값이 포함되어 있을 경우mle는data에서 이에 해당하는 행을 무시합니다.
데이터형: single | double
이름-값 인수
선택적 인수 쌍을 Name1=Value1,...,NameN=ValueN으로 지정합니다. 여기서 Name은 인수 이름이고 Value는 대응값입니다. 이름-값 인수는 다른 인수 뒤에 와야 하지만, 인수 쌍의 순서는 상관없습니다.
R2021a 이전 릴리스에서는 쉼표를 사용하여 각 이름과 값을 구분하고 Name을 따옴표로 묶으십시오.
예: 'Censoring',Cens,'Alpha',0.01,'Options',Opt는 mle가 배열 Cens로 지정된 중도절단된 데이터의 분포에 대한 모수를 추정하고 모수 추정값에 대한 99% 신뢰한계를 계산한 후 구조체 Opt로 지정된 알고리즘 제어 모수를 사용하도록 지시합니다.
내장 분포를 지정하는 옵션
모수를 추정할 분포 유형으로, 다음 표에 있는 값 중 하나로 지정됩니다.
Distribution 값 | 분포 유형 | 첫 번째 모수 | 두 번째 모수 | 세 번째 모수 | 네 번째 모수 |
|---|---|---|---|---|---|
'Bernoulli' | 베르누이 분포 | p: 각 시행에 대한 성공 확률 | N/A | N/A | N/A |
'Beta' | 베타 분포 | a: 첫 번째 형태 모수 | b: 두 번째 형태 모수 | N/A | N/A |
'Binomial' | 이항분포 | p: 각 시행에 대한 성공 확률 | N/A | N/A | N/A |
'BirnbaumSaunders' | Birnbaum-Saunders Distribution | β: 스케일 모수 | γ: 형태 모수 | N/A | N/A |
'Burr' | Burr Type XII Distribution | α: 스케일 모수 | c: 첫 번째 형태 모수 | k: 두 번째 형태 모수 | N/A |
'Discrete Uniform' 또는 'unid' | 균등분포(이산) | n: 관측 가능 최댓값 | N/A | N/A | N/A |
'Exponential' | 지수 분포 | μ: 평균 | N/A | N/A | N/A |
'Extreme Value' 또는 'ev' | Extreme Value Distribution | μ: 위치 모수 | σ: 스케일 모수 | N/A | N/A |
'Gamma' | 감마 분포 | a: 형태 모수 | b: 스케일 모수 | N/A | N/A |
'Generalized Extreme Value' 또는 'gev' | Generalized Extreme Value Distribution | k: 형태 모수 | σ: 스케일 모수 | μ: 위치 모수 | N/A |
'Generalized Pareto' 또는 'gp' | Generalized Pareto Distribution | k: 꼬리 인덱스(형태) 모수 | σ: 스케일 모수 | N/A | N/A |
'Geometric' | Geometric Distribution | p: 확률 모수 | N/A | N/A | N/A |
'Half Normal' 또는 'hn' | Half-Normal Distribution | σ: 스케일 모수 | N/A | N/A | N/A |
'InverseGaussian' | 역가우스 분포 | μ: 스케일 모수 | λ: 형태 모수 | N/A | N/A |
'Logistic' | 로지스틱 분포 | μ: 평균 | σ: 스케일 모수 | N/A | N/A |
'LogLogistic' | 로그-로지스틱 분포 | μ: 로그 값의 평균 | σ: 로그 값의 스케일 모수 | N/A | N/A |
'LogNormal' | 로그정규분포 | μ: 로그 값의 평균 | σ: 로그 값의 표준편차 | N/A | N/A |
'Nakagami' | 나카가미(Nakagami) 분포 | μ: 형태 모수 | ω: 스케일 모수 | N/A | N/A |
'Negative Binomial' 또는 'nbin' | Negative Binomial Distribution | r: 성공 횟수 | p: 단일 시행에서 성공할 확률 | N/A | N/A |
'Normal' | 정규분포 | μ: 평균 | σ: 표준편차 | N/A | N/A |
'Poisson' | 푸아송 분포 | λ: 평균 | N/A | N/A | N/A |
'Rayleigh' | 레일리(Rayleigh) 분포 | b: 스케일 모수 | N/A | N/A | N/A |
'Rician' | 라이시안(Rician) 분포 | s: 비중심성 모수 | σ: 스케일 모수 | N/A | N/A |
'Stable' | Stable Distribution | α: 첫 번째 형태 모수 | β: 두 번째 형태 모수 | γ: 스케일 모수 | δ: 위치 모수 |
'tLocationScale' | t Location-Scale Distribution | μ: 위치 모수 | σ: 스케일 모수 | ν: 형태 모수 | N/A |
'Uniform' | 균등분포(연속) | a: 하한 끝점(최솟값) | b: 상한 끝점(최댓값) | N/A | N/A |
'Weibull' 또는 'wbl' | 베이불(Weibull) 분포 | a: 스케일 모수 | b: 형태 모수 | N/A | N/A |
mle는 다음 분포 모수를 추정하지 않습니다.
표본 데이터가 절단되었거나 표본 데이터에 좌측 중도절단된 관측값 또는 구간 중도절단된 관측값이 포함된 경우, 버(Burr) 분포 및 안정분포에 대해 Start 이름-값 인수를 지정해야 합니다.
예: 'Distribution','Rician'
이항분포에 대해 data의 대응 요소에 사용할 시행 횟수로, data와 같은 행 개수의 스칼라 또는 벡터로 지정됩니다.
이 인수는 Distribution이 'Binomial'(이항분포)인 경우에만 필요합니다.
예: 'Ntrials',10
데이터형: single | double
일반화 파레토 분포에 사용할 위치(분계점) 모수로, 스칼라로 지정됩니다.
이 인수는 Distribution이 'Generalized Pareto'(일반화 파레토 분포)인 경우에만 유효합니다.
표본 데이터 data에 음이 아닌 값만 포함된 경우 디폴트 값은 0입니다. data에 음수 값이 포함된 경우에는 theta를 지정해야 합니다.
예: 'theta',1
데이터형: single | double
절반 정규분포에 사용할 위치 모수로, 스칼라로 지정됩니다.
이 인수는 Distribution이 'Half Normal'(절반 정규분포)인 경우에만 유효합니다.
표본 데이터 data에 음이 아닌 값만 포함된 경우 디폴트 값은 0입니다. data에 음수 값이 포함된 경우에는 mu를 지정해야 합니다.
예: 'mu',1
데이터형: single | double
사용자 지정 분포를 정의하는 옵션
사용자 지정 확률 밀도 함수(pdf)로, 함수 핸들로 지정되거나 함수 핸들 및 함수에 대한 추가 인수를 포함하는 셀형 배열로 지정됩니다.
이 사용자 지정 함수는 표본 데이터를 포함하는 벡터, 하나 이상의 개별 분포 모수, 셀형 배열로 전달되는 추가 인수를 입력 모수로 받습니다. 이 함수는 확률 밀도 값으로 구성된 벡터를 반환합니다.
예: 'pdf',@newpdf
데이터형: function_handle | cell
사용자 지정 누적 분포 함수(cdf)로, 함수 핸들로 지정되거나 함수 핸들 및 함수에 대한 추가 인수를 포함하는 셀형 배열로 지정됩니다.
이 사용자 지정 함수는 표본 데이터를 포함하는 벡터, 하나 이상의 개별 분포 모수, 셀형 배열로 전달되는 추가 인수를 입력 모수로 받습니다. 이 함수는 cdf 값으로 구성된 벡터를 반환합니다.
중도절단되거나 절단된 관측값에 대해 MLE를 계산하려면 cdf와 pdf를 모두 정의해야 합니다. 완전히 관측되고 절단되지 않은 관측값의 경우 mle는 cdf를 사용하지 않습니다. data 또는 Censoring을 사용하여 중도절단 정보를 지정하거나 TruncationBounds를 사용하여 절단 범위를 지정할 수 있습니다.
예: 'cdf',@newcdf
데이터형: function_handle | cell
사용자 지정 로그 확률 밀도 함수로, 함수 핸들로 지정되거나 함수 핸들 및 함수에 대한 추가 인수를 포함하는 셀형 배열로 지정됩니다.
이 사용자 지정 함수는 표본 데이터를 포함하는 벡터, 하나 이상의 개별 분포 모수, 셀형 배열로 전달되는 추가 인수를 입력 모수로 받습니다. 이 함수는 로그 확률 값으로 구성된 벡터를 반환합니다.
예: 'logpdf',@customlogpdf
데이터형: function_handle | cell
사용자 지정 로그 생존 함수로, 함수 핸들로 지정되거나 함수 핸들 및 함수에 대한 추가 인수를 포함하는 셀형 배열로 지정됩니다.
이 사용자 지정 함수는 표본 데이터를 포함하는 벡터, 하나 이상의 개별 분포 모수, 셀형 배열로 전달되는 추가 인수를 입력 모수로 받습니다. 이 함수는 로그 생존 확률 값으로 구성된 벡터를 반환합니다.
중도절단되거나 절단된 관측값에 대해 MLE를 계산하려면 logsf와 logpdf를 모두 정의해야 합니다. 완전히 관측되고 절단되지 않은 관측값의 경우 mle는 logsf를 사용하지 않습니다. data 또는 Censoring을 사용하여 중도절단 정보를 지정하거나 TruncationBounds를 사용하여 절단 범위를 지정할 수 있습니다.
예: 'logsf',@logsurvival
데이터형: function_handle | cell
사용자 지정 음의 로그 가능도 함수로, 함수 핸들로 지정되거나 함수 핸들 및 함수에 대한 추가 인수를 포함하는 셀형 배열로 지정됩니다.
이 사용자 지정 함수는 표에 나열된 순서대로 다음 입력 인수를 받습니다.
| 사용자 지정 함수의 입력 인수 | 설명 |
|---|---|
params | 분포 모수 값으로 구성된 벡터입니다. mle는 Start에 포함된 요소의 개수를 통해 모수의 개수를 감지합니다. |
data | 표본 데이터. data 값은 표본 데이터로 구성된 벡터나 표본 데이터 및 중도절단 정보로 구성된 2열 행렬로 지정됩니다. |
cens | 중도절단 정보로 구성된 논리형 벡터. Censoring 이름-값 인수를 사용하지 않는 경우에도 nloglf는 cens를 받아야 합니다. 이 경우 cens를 무시하도록 nloglf를 작성할 수 있습니다. |
freq | 데이터 도수로 구성된 정수 벡터. Frequency 이름-값 인수를 사용하지 않는 경우에도 nloglf는 freq를 받아야 합니다. 이 경우 freq를 무시하도록 nloglf를 작성할 수 있습니다. |
trunc | 절단 범위를 나타내는 요소 2개로 이루어진 숫자형 벡터. TruncationBounds 이름-값 인수를 사용하는 경우 nloglf는 trunc를 받아야 합니다. |
선택적으로, nloglf는 셀형 배열로 전달되는 추가 인수를 입력 모수로 받을 수 있습니다.
nloglf는 음의 스칼라 로그 가능도 값과 선택적으로 음의 로그 가능도 기울기 벡터를 반환합니다(Options 이름-값 인수의 GradObj 필드 참조).
예: 'nloglf',@negloglik
데이터형: function_handle | cell
기타 옵션
중도절단된 데이터 표시자로, 각각 완전히 관측된 관측값, 좌측 중도절단된 관측값 및 우측 중도절단된 관측값을 나타내는 0, –1 및 1로 구성된 벡터로 지정됩니다. Censoring 값의 각 요소는 data의 대응되는 관측값에 대한 중도절단 상태를 나타냅니다. Censoring 값은 data와 크기가 같아야 합니다. 디폴트 값은 0으로 구성된 벡터로, 모든 관측값이 완전히 관측됨을 나타냅니다.
이 인수를 사용하여 구간 중도절단된 관측값을 지정할 수 없습니다. 표본 데이터에 구간 중도절단된 관측값이 포함된 경우 2열 행렬을 사용하여 data를 지정하십시오. data가 2열 행렬인 경우 mle는 Censoring 값을 무시합니다.
mle는 다음과 같은 내장 분포 및 사용자 지정 분포에 대해 중도절단을 지원합니다.
Distribution 값 | 분포 유형 |
|---|---|
'BirnbaumSaunders' | 번바움-손더스(Birnbaum-Saunders) |
'Burr' | 버(Burr) 타입 XII |
'Exponential' | 지수 |
'Extreme Value' 또는 'ev' | 극값 |
'Gamma' | 감마 |
'InverseGaussian' | 역가우스 |
'Logistic' | 로지스틱 |
'LogLogistic' | 로그-로지스틱 |
'LogNormal' | 로그정규 |
'Nakagami' | 나카가미(Nakagami) |
'Normal' | 정규분포 |
'Rician' | 라이시안(Rician) |
'tLocationScale' | t 위치-척도 |
'Weibull' 또는 'wbl' | 베이불(Weibull) |
사용자 지정 분포의 경우, pdf 및 cdf, logpdf 및 logsf, 또는 nloglf를 사용하여 분포를 정의해야 합니다.
mle는 중도절단 벡터에 포함된 NaN 값을 무시합니다. 또한, data 또는 도수 벡터(Frequency)에 NaN 값이 하나라도 포함되어 있으면 mle가 중도절단 벡터에서 이에 대응되는 값을 무시합니다.
예: 'Censoring',censored. 여기서 censored는 중도절단 정보를 포함하는 벡터입니다.
데이터형: logical | single | double
절단 범위로, 두 요소로 구성된 벡터로 지정됩니다.
mle는 다음과 같은 내장 분포 및 사용자 지정 분포에 대해 절단된 관측값을 지원합니다.
Distribution 값 | 분포 유형 |
|---|---|
'Beta' | 베타 |
'BirnbaumSaunders' | 번바움-손더스(Birnbaum-Saunders) |
'Burr' | 버(Burr) |
'Exponential' | 지수 |
'Extreme Value' 또는 'ev' | 극값 |
'Gamma' | 감마 |
'Generalized Extreme Value' 또는 'gev' | 일반화 극값 |
'Generalized Pareto' 또는 'gp' | 일반화 파레토 |
'Half Normal' 또는 'hn' | 절반 정규 |
'InverseGaussian' | 역가우스 |
'Logistic' | 로지스틱 |
'LogLogistic' | 로그-로지스틱 |
'LogNormal' | 로그정규 |
'Nakagami' | 나카가미(Nakagami) |
'Normal' | 정규분포 |
'Poisson' | 푸아송 |
'Rayleigh' | 레일리(Rayleigh) |
'Rician' | 라이시안(Rician) |
'Stable' | 안정 |
'tLocationScale' | t 위치-척도 |
'Weibull' 또는 'wbl' | 베이불(Weibull) |
사용자 지정 분포의 경우, pdf 및 cdf, logpdf 및 logsf, 또는 nloglf를 사용하여 분포를 정의해야 합니다.
예: 'TruncationBounds',[0,10]
데이터형: single | double
관측값의 도수로, data와 동일한 행 개수를 갖는, 음이 아닌 정수 도수로 구성된 벡터로 지정됩니다. Frequency 값의 j번째 요소는 data의 j번째 행이 관측된 횟수를 알려줍니다. 디폴트 값은 1로 구성된 벡터로, data의 행당 하나의 관측값이 있음을 나타냅니다.
mle는 이러한 도수 벡터에 포함된 NaN 값을 모두 무시합니다. 또한, data 또는 중도절단 벡터(Censoring)에 NaN 값이 하나라도 포함되어 있으면 mle가 도수 벡터에서 이에 대응되는 값을 무시합니다.
예: 'Frequency',freq. 여기서 freq는 관측값의 도수를 포함하는 벡터입니다.
데이터형: single | double
모수 추정값의 신뢰구간 pci에 대한 유의수준으로, (0,1) 범위의 스칼라로 지정됩니다. pci의 신뢰수준은 100(1–Alpha)%입니다. 디폴트 값은 95% 신뢰도에 대해 0.05입니다.
예: 'Alpha',0.01은 신뢰수준을 99%로 지정합니다.
데이터형: single | double
반복 알고리즘에 사용할 옵션으로, statset으로 반환되는 구조체로 지정됩니다.
이 인수를 사용하여 최대가능도 최적화에 대한 세부 정보를 제어할 수 있습니다. 이 인수는 다음과 같은 경우에만 유효합니다.
표본 데이터가 절단되었습니다.
표본 데이터에 좌측 중도절단된 관측값이나 구간 중도절단된 관측값이 포함되어 있습니다.
사용자 지정 분포를 피팅합니다.
mle 함수는 최적화에 대한 다음과 같은 statset 옵션을 해석합니다.
| 필드 이름 | 설명 | 디폴트 값 |
|---|---|---|
GradObj |
| 'off' |
DerivStep | 상대 오차로,
| eps^(1/3) |
FunValCheck |
분포 함수를 정의할 때 적절한 오류 검사를 포함시키지 않으면 시작점을 잘못 선택하는 경우 함수가 | 'on' |
TolBnd |
| 1e-6 |
TolFun | 함수 값에 대한 종료 허용오차로, 양의 스칼라로 지정됩니다. | 1e-6 |
TolX | 모수에 대한 종료 허용오차로, 양의 스칼라로 지정됩니다. | 1e-6 |
MaxFunEvals | 허용되는 함수 실행의 최대 횟수로, 양의 정수로 지정됩니다. | 400 |
MaxIter | 허용되는 최대 반복 횟수로, 양의 정수로 지정됩니다. | 200 |
Display | 표시 수준으로,
| 'off' |
Options 이름-값 인수에 대한 예제는 유한 지지 범위를 가진 분포에 대한 MLE 구하기 및Three-Parameter Weibull Distribution 항목을 참조하십시오.
자세한 내용은 fminsearch 및 fmincon (Optimization Toolbox)의 options 입력 인수를 참조하십시오.
예: 'Options',statset('FunValCheck','off')
데이터형: struct
버(Burr) 분포, 안정분포, 사용자 지정 분포에 대한 초기 모수 값으로, 행 벡터로 지정됩니다. Start 값의 길이는 mle로 추정된 모수의 개수와 같아야 합니다.
표본 데이터가 절단되었거나 표본 데이터에 좌측 중도절단된 관측값 또는 구간 중도절단된 관측값이 포함된 경우, 버 분포 및 안정분포에 대해 Start 인수가 필요합니다. 이 인수는 사용자 지정 분포를 피팅하는 경우, 즉 pdf, logpdf 또는 nloglf 이름-값 인수를 사용하는 경우 항상 필요합니다. 이 외의 경우, mle는 초기값을 찾거나 초기값 없이 MLE를 계산할 수 있습니다.
예: 0.05
예: [100,2]
데이터형: single | double
분포 모수에 대한 하한으로, Start와 길이가 같은 행 벡터로 지정됩니다.
이 인수는 다음과 같은 경우에만 유효합니다.
표본 데이터가 절단되었습니다.
표본 데이터에 좌측 중도절단된 관측값이나 구간 중도절단된 관측값이 포함되어 있습니다.
사용자 지정 분포를 피팅합니다.
예: 'Lowerbound',0
데이터형: single | double
분포 모수에 대한 상한으로, Start와 길이가 같은 행 벡터로 지정됩니다.
이 인수는 다음과 같은 경우에만 유효합니다.
표본 데이터가 절단되었습니다.
표본 데이터에 좌측 중도절단된 관측값이나 구간 중도절단된 관측값이 포함되어 있습니다.
사용자 지정 분포를 피팅합니다.
예: 'Upperbound',1
데이터형: single | double
mle가 가능도를 최대화하는 데 사용하는 최적화 함수로, 'fminsearch' 또는 'fmincon'으로 지정됩니다. 'fmincon' 옵션을 사용하려면 Optimization Toolbox™가 필요합니다.
표본 데이터가 절단되었습니다.
표본 데이터에 좌측 중도절단된 관측값이나 구간 중도절단된 관측값이 포함되어 있습니다.
사용자 지정 분포를 피팅합니다.
예: 'Optimfun','fmincon'
출력 인수
모수 추정값으로, 행 벡터로 반환됩니다. 내장 분포의 모수 추정값에 대한 설명은 Distribution 항목을 참조하십시오.
모수 추정값의 신뢰구간으로, 2×k 행렬로 반환됩니다. 여기서 k는 mle로 추정된 모수의 개수입니다. pci의 첫 번째 행과 두 번째 행은 각각 신뢰 하한과 신뢰 상한을 보여줍니다.
Alpha 이름-값 인수를 사용하여 신뢰구간에 대해 유의수준을 지정할 수 있습니다.
세부 정보
mle 함수는 좌측 중도절단된 관측값, 우측 중도절단된 관측값 및 구간 중도절단된 관측값을 지원합니다.
시간
t에서 좌측 중도절단된 관측값 — 이벤트가 시간t이전에 발생했으며 정확한 이벤트 시간은 알 수 없습니다.시간
t에서 우측 중도절단된 관측값 — 이벤트가 시간t이후에 발생했으며 정확한 이벤트 시간은 알 수 없습니다.구간
[t1,t2]내에서 구간 중도절단된 관측값 — 이벤트가 시간t1이후 및 시간t2이전에 발생했으며 정확한 이벤트 시간은 알 수 없습니다.
양측 중도절단된 데이터는 좌측 중도절단된 관측값과 우측 중도절단된 관측값을 모두 포함합니다.
생존 함수는 시간에 대한 함수로 표현되는 생존 확률입니다. 이는 생존자 함수(Survivor Function)라고도 합니다.
이 생존 함수는 개별 항목의 생존 시간이 특정 값을 초과할 확률을 제공합니다. 누적 분포 함수 F(t)는 생존 시간이 주어진 특정 시점 t보다 작거나 같을 확률이므로 연속 분포에 대한 생존 함수 S(t)는 누적 분포 함수의 보수, 즉 S(t) = 1 – F(t)입니다.
팁
사용자 지정 분포 함수를 제공하거나 좌측 중도절단된 관측값, 양측 중도절단된 관측값, 구간 중도절단된 관측값 또는 절단된 관측값에 대해 내장 분포를 사용하는 경우
mle는 반복적 최대화 알고리즘을 사용하여 모수 추정값을 계산합니다. 일부 모델 및 데이터에서 시작점(Start)을 잘못 선택하는 경우mle가 전역 최댓값이 아닌 국소 최적해로 수렴하거나 전혀 수렴하지 않을 수 있습니다. 심지어 로그 가능도가 전역 최댓값 주변에서 잘 동작하는 경우에도 시작점 선택이 알고리즘 수렴에 중요한 요소가 되는 경우가 자주 있습니다. 특히, 초기 모수 값이 MLE에서 멀리 떨어진 경우, 분포 함수의 언더플로로 인해 무한대의 로그 가능도가 초래될 수 있습니다.
알고리즘
mle함수는 음의 로그 가능도 함수를 최소화하거나(즉, 로그 가능도 함수를 최대화) 가능한 경우 닫힌 형식 해를 사용하여 MLE를 구합니다. 목적 함수는 분포 모수(θ)가 주어졌을 때 표본 데이터(X) 확률의 곱의 음의 로그 값입니다.확률 함수 P는 각 관측값에 대한 중도절단 정보에 따라 달라집니다.
완전히 관측된 관측값 — P(x|θ) = f(x). 여기서 f는 모수 θ를 갖는 확률 밀도 함수(pdf)입니다.
좌측 중도절단된 관측값 — P(x|θ) = F(x). 여기서 F는 모수 θ를 갖는 누적 분포 함수(cdf)입니다.
우측 중도절단된 관측값 — P(x|θ) = 1 – F(x).
xL과 xU 사이의 구간 중도절단된 관측값 — P(x|θ) = F(xU) – F(xL).
절단된 데이터의 경우,
mle는 모든 확률이 절단 범위 [L,U] 내에 있도록 분포 함수의 크기를 스케일링합니다.표본 데이터가 절단되지 않았고 표본 데이터에 좌측 중도절단된 관측값 또는 구간 중도절단된 관측값이 포함되지 않은 경우
mle함수는 가능한 경우 정확한 방법을 사용하여 신뢰구간pci를 계산합니다. 그렇지 않은 경우 함수는 왈드 방법을 사용합니다. 정확한 방법은 이항분포, 이산 균등분포, 지수 분포, 정규분포, 로그정규분포, 푸아송 분포, 레일리 분포, 연속 균등분포에 사용할 수 있습니다.
확장 기능
사용법 관련 참고 및 제한 사항:
이름-값 인수
Distribution을'Rician'또는'Stable'로 지정할 수 없습니다.pdf및cdf,logpdf및logsf, 또는nloglf이름-값 인수를 사용하여 사용자 지정 분포를 피팅하는 경우 사용자 지정 분포 함수는 GPU 배열을 지원해야 합니다.
자세한 내용은 GPU에서 MATLAB 함수 실행하기 (Parallel Computing Toolbox) 항목을 참조하십시오.
버전 내역
R2006a 이전에 개발됨
MATLAB Command
You clicked a link that corresponds to this MATLAB command:
Run the command by entering it in the MATLAB Command Window. Web browsers do not support MATLAB commands.
웹사이트 선택
번역된 콘텐츠를 보고 지역별 이벤트와 혜택을 살펴보려면 웹사이트를 선택하십시오. 현재 계신 지역에 따라 다음 웹사이트를 권장합니다:
또한 다음 목록에서 웹사이트를 선택하실 수도 있습니다.
사이트 성능 최적화 방법
최고의 사이트 성능을 위해 중국 사이트(중국어 또는 영어)를 선택하십시오. 현재 계신 지역에서는 다른 국가의 MathWorks 사이트 방문이 최적화되지 않았습니다.
미주
- América Latina (Español)
- Canada (English)
- United States (English)
유럽
- Belgium (English)
- Denmark (English)
- Deutschland (Deutsch)
- España (Español)
- Finland (English)
- France (Français)
- Ireland (English)
- Italia (Italiano)
- Luxembourg (English)
- Netherlands (English)
- Norway (English)
- Österreich (Deutsch)
- Portugal (English)
- Sweden (English)
- Switzerland
- United Kingdom (English)