linkage
계층적 병합 군집 트리
구문
설명
예제
입력 인수
출력 인수
세부 정보
팁
y가 거리 행렬의 벡터 표현인 경우linkage(y)계산이 느려질 수 있습니다.'centroid','median','ward'방법에 대해linkage는y가 유클리드 거리인지 여부를 확인합니다.y대신X를 전달하여 시간이 많이 걸리는 이 확인 작업을 피할 수 있습니다.'centroid'방법과'median'방법은 단조적(Monotonic)이지 않은 군집 트리를 생성할 수 있습니다. 이 결과는 두 군집 r과 s의 합집합에서 세 번째 군집까지의 거리가 r과 s 간의 거리보다 작을 경우에 발생합니다. 이 경우, 디폴트 방향으로 그린 덴드로그램에서 리프로부터 루트 노드까지의 경로는 아래쪽 방향의 일부 단계를 지나게 됩니다. 이 결과를 방지하려면 다른 방법을 사용하십시오. 다음 Figure는 비단조적 군집 트리를 보여줍니다.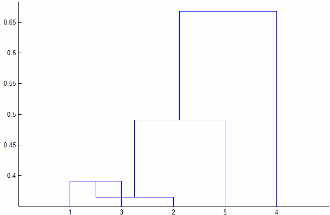
이 경우, 군집 1과 군집 3이 새 군집으로 결합되며, 이 새 군집과 군집 2 간의 거리는 군집 1과 군집 3 간의 거리보다 작습니다. 그 결과 비단조적 트리가 생성됩니다.
트리를 표시하기 위한
dendrogram, 점을 군집에 할당하기 위한cluster, 일치하지 않는 측정값을 계산하기 위한inconsistent, 코페네틱 상관 계수(Cophenetic Correlation Coefficient)를 계산하기 위한cophenet를 비롯한 다른 함수에 출력값Z를 제공할 수 있습니다.
버전 내역
R2006a 이전에 개발됨
참고 항목
cluster | clusterdata | cophenet | dendrogram | inconsistent | kmeans | pdist | silhouette | squareform