세포 분할에 사용할 사전 훈련된 Cellpose 모델 선택하기
이 예제에서는 사전 훈련된 Cellpose 모델을 사용하여 현미경 영상에서 세포를 분할하는 방법을 보여줍니다.
Cellpose Library[1],[2]는 현미경 영상에서 세포를 분할하는 데 사용할 수 있는 여러 사전 훈련된 모델을 제공합니다. 각 모델과 그 훈련 데이터에 대한 자세한 내용은 Cellpose Library Documentation의 Models 페이지를 참조하십시오. 이 예제에서는 사전 훈련된 Cellpose 모델 3개를 사용하여 영상을 분할하고 결과를 시각적으로 비교합니다. 이 방식을 통해 사용자 고유의 영상에 가장 적합한 사전 훈련된 모델을 찾을 수 있습니다. 사전 훈련된 모델 중에 영상 데이터 세트에서 잘 동작하는 것이 없는 경우 전이 학습을 통해 사용자 지정 Cellpose 모델을 훈련시킬 수 있습니다. 훈련에 대한 자세한 내용은 Train Custom Cellpose Model 예제를 참조하십시오.
이 예제를 수행하려면 Medical Imaging Toolbox™ Interface for Cellpose Library가 필요합니다. 애드온 탐색기에서 Medical Imaging Toolbox Interface for Cellpose Library를 설치할 수 있습니다. 애드온 설치에 대한 자세한 내용은 애드온을 받고 관리하기 항목을 참조하십시오. Medical Imaging Toolbox Interface for Cellpose Library를 사용하려면 Deep Learning Toolbox™와 Computer Vision Toolbox™가 필요합니다.
영상 불러오기
영상을 읽어 들여 표시합니다. 이 예제에서는 메모리에 담을 수 있을 정도로 작은 단일채널 회색조 영상을 사용합니다. 사용자 고유의 영상으로 작업하는 경우 다음 팁을 참조하는 게 좋습니다.
Cellpose 모델에는 입력값으로 2차원 명암 영상이 필요합니다. RGB 또는 다중채널 영상이 있는 경우
im2gray함수를 사용하여 회색조로 변환하거나imsplit함수를 사용하여 특정 색 채널을 추출할 수 있습니다. 일반적으로 세포 경계의 가장자리가 가장 강한 채널을 선택하십시오. 영상에 핵 염색을 위한 별도의 채널이 포함되어 있는 경우segmentCells2D함수의AuxiliaryChannel이름-값 인수를 사용하여 분할 중에 핵 영상을 지정하는 것이 좋습니다.분할할 영상이 많은 경우, 대표적인 영상이나 그러한 영상들 중 일부를 선택하십시오. 전체 데이터 세트에서 레이블을 지정할 유형과 형태의 세포가 포함된 영상을 사용하십시오.
전체 슬라이드 현미경 영상처럼 크기가 큰 영상으로 작업하는 경우 관심 있는 대표 영역을 추출하십시오. 전체 영상에서 레이블을 지정할 세포 유형이 포함된 영역을 선택하십시오.
blockedImage객체와getRegion객체 함수를 사용하여 크기가 큰 현미경 영상에서 영역을 추출하는 예제는 부분 영상 또는 낮은 해상도를 사용하여 효율적으로 블록 형식 영상 처리하기 항목을 참조하십시오.
img = imread("AT3_1m4_01.tif");
imshow(img)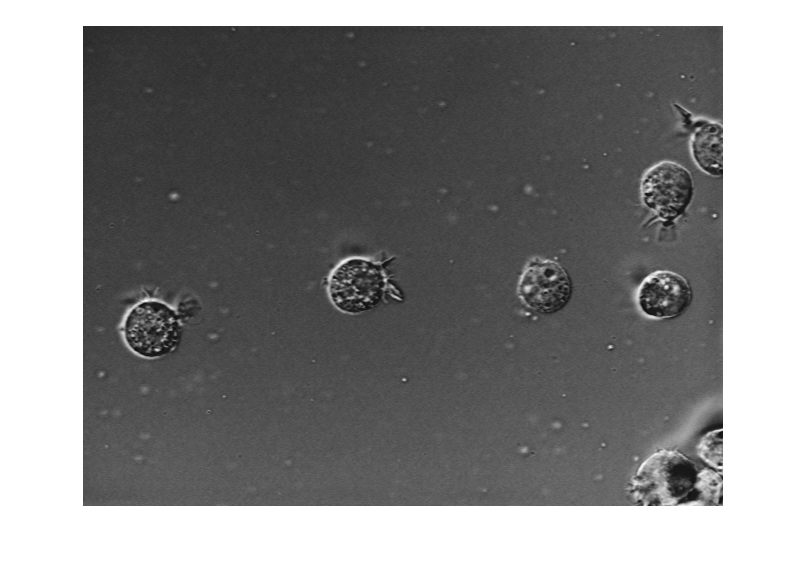
평균 세포 지름 측정하기
정확한 결과를 얻기 위해 세포의 대략적인 지름(단위: 픽셀)을 지정합니다. 이 예제에서는 지름이 56픽셀이라고 가정합니다.
averageCellDiameter = 56;
또는 영상 뷰어 앱에서 대략적인 지름을 측정할 수 있습니다. 다음 명령을 입력하여 이 앱을 엽니다.
imageViewer(img)
앱 툴스트립의 뷰어 탭에서 거리 측정을 클릭합니다. 메인 표시 창에서 측정하려는 세포 경계의 한 점을 클릭하고 끌어서 세포에 가로로 선을 그립니다. 버튼을 놓으면 거리 측정값(단위: 픽셀)과 함께 선에 레이블이 지정됩니다. 선의 끝부분 또는 중간 부분을 각각 끌어서 끝점의 위치를 바꾸거나 전체 선을 옮길 수 있습니다. 다음 영상에는 앱에서 측정한 여러 거리 측정값의 예가 나와 있습니다.

Cellpose 모델을 사용하여 영상 분할하기
이 섹션에서는 사전 훈련된 Cellpose Library 모델 3개(cyto, cyto2, nuclei)에서 얻은 결과를 비교합니다.
Cellpose 모델 구성하기
각 모델에 대한 cellpose 객체를 만들고 각 객체의 Model 속성을 지정합니다. 사용자가 주어진 모델에 대한 객체를 처음 생성할 경우 MATLAB®이 Cellpose Library에서 모델을 다운로드하고 로컬 복사본을 저장합니다. 기본적으로 MATLAB은 userpath 함수에서 반환된 폴더 내 cellposeModels라는 폴더에 모델을 저장합니다. 저장 위치는 객체를 만들 때 ModelFolder 속성을 지정하여 변경할 수 있습니다.
cpCyto = cellpose(Model="cyto"); cpCyto2 = cellpose(Model="cyto2"); cpNuclei = cellpose(Model="nuclei");
세포 분할 레이블 예측하기
세 가지 각 모델을 사용하여 segmentCells2D 객체 함수로 영상을 분할합니다.
먼저, 세포 임계값과 흐름 오차 임계값의 값을 지정합니다. 일반적으로 세포 임계값의 범위는 -6~6이고, 흐름 오차 임계값의 일반적 범위는 0.1~3입니다. 이 예제에서는 검출 수를 더 많이 늘릴 수 있도록 다음 값을 설정합니다.
cellThreshold = -6; flowThreshold = 3;
각 모델의 레이블을 예측합니다. segmentCells2D 객체 함수는 2차원 레이블 영상을 반환하며, 이때 모델에서 검출된 각 세포는 서로 다른 숫자형 픽셀 값을 갖습니다.
labels1 = segmentCells2D(cpCyto,img, ... ImageCellDiameter=averageCellDiameter, ... CellThreshold=cellThreshold, ... FlowErrorThreshold=flowThreshold); labels2 = segmentCells2D(cpCyto2,img, ... ImageCellDiameter=averageCellDiameter, ... CellThreshold=cellThreshold, ... FlowErrorThreshold=flowThreshold); labels3 = segmentCells2D(cpNuclei,img, ... ImageCellDiameter=averageCellDiameter, ... CellThreshold=cellThreshold, ... FlowErrorThreshold=flowThreshold);
결과 비교
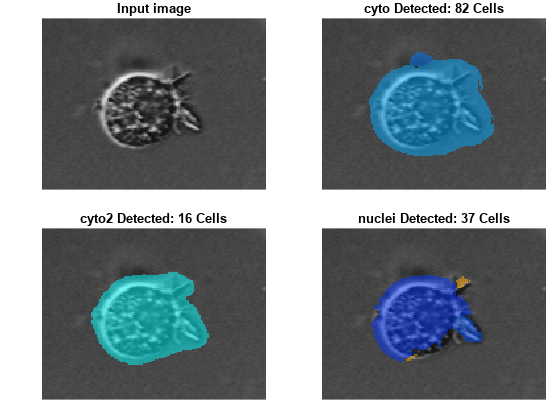
입력 영상과 각 모델의 결과를 시각화합니다. 각 영상의 제목에는 모델 이름과 검출된 세포 개수가 포함되어 있습니다.
figure tiledlayout(2,2,TileSpacing="tight",Padding="tight") nexttile h1 = imshow(img); title("Input image") nexttile h2 = imshow(labeloverlay(img,labels1)); nl = numel(unique(labels1(:))) - 1; title(cpCyto.Model + " Detected: " + nl + " Cells") nexttile h3 = imshow(labeloverlay(img,labels2)); nl = numel(unique(labels2(:))) - 1; title(cpCyto2.Model + " Detected: " + nl + " Cells") nexttile h4 = imshow(labeloverlay(img,labels3)); nl = numel(unique(labels3(:))) - 1; title(cpNuclei.Model + " Detected: " + nl + " Cells") linkaxes([h1.Parent h2.Parent h3.Parent h4.Parent])

각 영상을 확대하여 각 모델에서 얻은 단일 세포에 대한 결과를 비교합니다. 일반적으로 사용자가 선택하고자 하는 모델은 실제 세포 하나를 레이블 영상의 세포 하나로 검출해 낼 정도로 올바른 세포 수를 정확하게 감지하는 모델입니다. 확대된 영상에 따르면 이 영상군에는 cyto2 모델이 가장 적합합니다.
xlim([200 370]) ylim([190 320])
모델을 선택했으면 세포 임계값과 흐름 오차 임계값과 같은 파라미터를 조정하여 경계의 정확도를 개선할 수 있습니다. 자세한 내용은 Refine Cellpose Segmentation by Tuning Model Parameters 예제를 참조하십시오.
참고 문헌
[1] Stringer, Carsen, Tim Wang, Michalis Michaelos, and Marius Pachitariu. “Cellpose: A Generalist Algorithm for Cellular Segmentation.” Nature Methods 18, no. 1 (January 2021): 100–106. https://doi.org/10.1038/s41592-020-01018-x.
[2] Pachitariu, Marius, and Carsen Stringer. “Cellpose 2.0: How to Train Your Own Model.” Nature Methods 19, no. 12 (December 2022): 1634–41. https://doi.org/10.1038/s41592-022-01663-4.
참고 항목
cellpose | segmentCells2D | trainCellpose | segmentCells3D | downloadCellposeModels
도움말 항목
- Refine Cellpose Segmentation by Tuning Model Parameters
- Train Custom Cellpose Model
- Detect Nuclei in Large Whole Slide Images Using Cellpose