fitgmdist
가우스 혼합 모델을 데이터에 피팅
설명
예제
두 개의 혼합된 이변량 가우스 분포에서 데이터를 생성합니다.
mu1 = [1 2];
Sigma1 = [2 0; 0 0.5];
mu2 = [-3 -5];
Sigma2 = [1 0;0 1];
rng(1); % For reproducibility
X = [mvnrnd(mu1,Sigma1,1000); mvnrnd(mu2,Sigma2,1000)];가우스 혼합 모델을 피팅합니다. 두 개의 성분이 있다고 지정합니다.
GMModel = fitgmdist(X,2);
피팅된 가우스 혼합 모델 등고선에 데이터를 플로팅합니다.
figure y = [zeros(1000,1);ones(1000,1)]; h = gscatter(X(:,1),X(:,2),y); hold on gmPDF = @(x,y) arrayfun(@(x0,y0) pdf(GMModel,[x0 y0]),x,y); g = gca; fcontour(gmPDF,[g.XLim g.YLim]) title('{\bf Scatter Plot and Fitted Gaussian Mixture Contours}') legend(h,'Model 0','Model1') hold off

두 개의 혼합된 이변량 가우스 분포에서 데이터를 생성합니다. 첫 번째 예측 변수와 두 번째 예측 변수의 합인 세 번째 예측 변수를 생성합니다.
mu1 = [1 2];
Sigma1 = [1 0; 0 1];
mu2 = [3 4];
Sigma2 = [0.5 0; 0 0.5];
rng(3); % For reproducibility
X1 = [mvnrnd(mu1,Sigma1,100);mvnrnd(mu2,Sigma2,100)];
X = [X1,X1(:,1)+X1(:,2)];X의 열은 선형 종속입니다. 이는 조건이 나쁜 공분산 추정값을 초래할 수 있습니다.
가우스 혼합 모델을 데이터에 피팅합니다. try/catch 문을 사용하면 오류 메시지를 관리하는 데 도움이 될 수 있습니다.
rng(1); % Reset seed for common start values try GMModel = fitgmdist(X,2) catch exception disp('There was an error fitting the Gaussian mixture model') error = exception.message end
There was an error fitting the Gaussian mixture model
error = 'Ill-conditioned covariance created at iteration 3.'
공분산 추정값은 조건이 나쁩니다. 따라서, 최적화가 중지되고 오류가 표시됩니다.
가우스 혼합 모델을 다시 피팅하되, 이번에는 정규화를 사용합니다.
rng(3); % Reset seed for common start values GMModel = fitgmdist(X,2,'RegularizationValue',0.1)
GMModel = Gaussian mixture distribution with 2 components in 3 dimensions Component 1: Mixing proportion: 0.536725 Mean: 2.8831 3.9506 6.8338 Component 2: Mixing proportion: 0.463275 Mean: 0.8813 1.9758 2.8571
이 경우, 알고리즘이 정규화로 인해 해로 수렴합니다.
가우스 혼합 모델을 사용하려면 데이터에 피팅하기 전에 성분의 개수를 지정해야 합니다. 많은 응용 사례의 경우 적합한 성분 개수를 파악하는 것이 어려울 수 있습니다. 이 예제에서는 주성분 분석을 사용하여 데이터를 탐색하고 성분의 개수에 대한 초기 추측값을 구하는 방법을 보여줍니다.
피셔(Fisher)의 붓꽃 데이터 세트를 불러옵니다.
load fisheriris
classes = unique(species)classes = 3×1 cell
{'setosa' }
{'versicolor'}
{'virginica' }
데이터 세트에는 붓꽃 종에 대한 세 개 클래스가 있습니다. 분석은 마치 이런 사실을 모르는 것처럼 진행됩니다.
시각화를 위해 데이터 차원을 2차원으로 줄이려면 주성분 분석을 사용하십시오.
[~,score] = pca(meas,'NumComponents',2);각각 1개, 2개, 3개 성분을 지정하여 세 개의 가우스 혼합 모델을 데이터에 피팅합니다. 최적화 반복 횟수를 1000으로 늘립니다. 점 표기법을 사용하여 최종 모수 추정값을 저장합니다. 기본적으로, 소프트웨어는 각 성분에 대해 완전 공분산과 여러 다른 공분산을 피팅합니다.
GMModels = cell(3,1); % Preallocation options = statset('MaxIter',1000); rng(1); % For reproducibility for j = 1:3 GMModels{j} = fitgmdist(score,j,'Options',options); fprintf('\n GM Mean for %i Component(s)\n',j) Mu = GMModels{j}.mu end
GM Mean for 1 Component(s)
Mu = 1×2
10-15 ×
0.9607 -0.4371
GM Mean for 2 Component(s)
Mu = 2×2
1.3212 -0.0954
-2.6424 0.1909
GM Mean for 3 Component(s)
Mu = 3×2
0.4856 -0.1287
1.4484 -0.0904
-2.6424 0.1909
GMModels는 세 가지 피팅된 gmdistribution 모델을 포함하는 셀형 배열입니다. 세 가지 성분 모델의 평균이 서로 다릅니다. 이는 모델이 세 가지 붓꽃 종을 구분한다는 것을 나타냅니다.
피팅된 가우스 혼합 모델 등고선에 점수를 플로팅합니다. 데이터 세트가 레이블을 포함하므로 성분의 실제 개수를 구분할 수 있도록 gscatter를 사용하십시오.
figure for j = 1:3 subplot(2,2,j) h1 = gscatter(score(:,1),score(:,2),species); h = gca; hold on gmPDF = @(x,y) arrayfun(@(x0,y0) pdf(GMModels{j},[x0 y0]),x,y); fcontour(gmPDF,[h.XLim h.YLim],'MeshDensity',100) title(sprintf('GM Model - %i Component(s)',j)); xlabel('1st principal component'); ylabel('2nd principal component'); if(j ~= 3) legend off; end hold off end g = legend(h1); g.Position = [0.7 0.25 0.1 0.1];
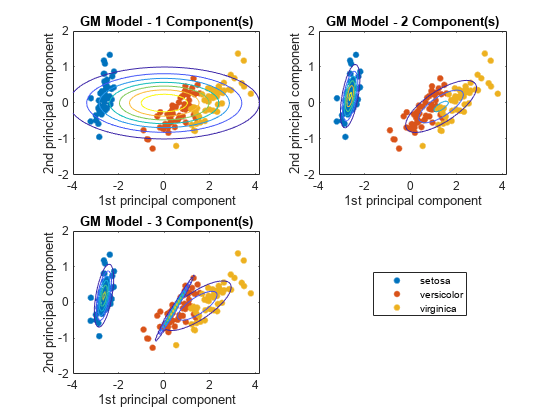
세 개 성분을 갖는 가우스 혼합 모델은 PCA와 함께 사용할 경우 세 가지 붓꽃 종을 구분하는 것처럼 보입니다.
가우스 혼합 모델에 적합한 성분 개수를 선택하는 데 도움이 되는 다른 옵션을 사용할 수 있습니다. 예를 들면 다음과 같습니다.
정보 기준(예: AIC 또는 BIC)을 사용하여 다양한 성분 개수를 갖는 여러 모델을 비교합니다.
Calinski-Harabasz 기준과 갭 통계량 또는 기타 기준을 지원하는
evalclusters를 사용하여 군집 개수를 추정합니다.
가우스 혼합 모델을 사용하려면 데이터에 피팅하기 전에 성분의 개수를 지정해야 합니다. 많은 응용 사례의 경우 적합한 성분 개수를 파악하는 것이 어려울 수 있습니다. 이 예제에서는 다양한 개수의 성분에서 최적의 피팅 가우스 혼합 모델을 선택하는 데 도움이 되도록 AIC 피팅 통계량을 사용합니다.
두 개의 혼합된 이변량 가우스 분포에서 데이터를 생성합니다.
mu1 = [1 1]; Sigma1 = [0.5 0; 0 0.5]; mu2 = [2 4]; Sigma2 = [0.2 0; 0 0.2]; rng(1); X = [mvnrnd(mu1,Sigma1,1000);mvnrnd(mu2,Sigma2,1000)]; plot(X(:,1),X(:,2),'ko') title('Scatter Plot') xlim([min(X(:)) max(X(:))]) % Make axes have the same scale ylim([min(X(:)) max(X(:))])
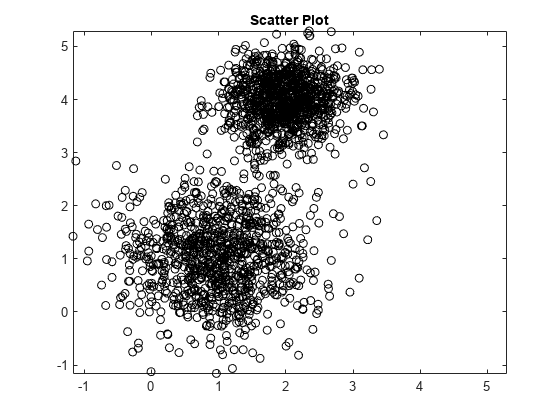
기본 모수 값을 알지 못한다고 가정해도 산점도 플롯을 보면 다음을 알 수 있습니다.
두 개 성분이 있습니다.
군집 간 분산이 다릅니다.
군집 내 분산이 같습니다.
군집 내에 공분산이 없습니다.
두 개 성분을 갖는 가우스 혼합 모델을 피팅합니다. 산점도 플롯을 관찰한 결과를 기반으로 하여 공분산 행렬이 대각 행렬임을 지정하십시오. statset 구조체를 Options 이름-값 쌍의 인수 값으로 전달하여 최종 반복 및 로그 가능도 통계량을 명령 창에 출력합니다.
options = statset('Display','final'); GMModel = fitgmdist(X,2,'CovarianceType','diagonal','Options',options);
11 iterations, log-likelihood = -4787.38
GMModel은 피팅된 gmdistribution 모델입니다.
다양한 개수의 성분에 대해 AIC를 검토합니다.
AIC = zeros(1,4); GMModels = cell(1,4); options = statset('MaxIter',500); for k = 1:4 GMModels{k} = fitgmdist(X,k,'Options',options,'CovarianceType','diagonal'); AIC(k)= GMModels{k}.AIC; end [minAIC,numComponents] = min(AIC); numComponents
numComponents = 2
BestModel = GMModels{numComponents}BestModel = Gaussian mixture distribution with 2 components in 2 dimensions Component 1: Mixing proportion: 0.501719 Mean: 1.9824 4.0013 Component 2: Mixing proportion: 0.498281 Mean: 0.9880 1.0511
두 개 성분을 갖는 가우스 혼합 모델을 피팅할 때 가장 작은 AIC가 발생합니다.
가우스 혼합 모델 모수 추정값은 초기값에 따라 달라질 수 있습니다. 이 예제에서는 fitgmdist를 사용하여 가우스 혼합 모델을 피팅할 때 초기값을 제어하는 방법을 보여줍니다.
피셔(Fisher)의 붓꽃 데이터 세트를 불러옵니다. 꽃잎 길이와 너비를 예측 변수로 사용합니다.
load fisheriris
X = meas(:,3:4);디폴트 초기값을 사용하여 가우스 혼합 모델을 데이터에 피팅합니다. 세 가지 벚꽃 종이 있으므로 k = 3으로 성분 개수를 지정합니다.
rng(10); % For reproducibility
GMModel1 = fitgmdist(X,3);기본적으로 소프트웨어는 다음을 수행합니다.
초기화를 위한 k-평균++ 알고리즘을 구현하여 k = 3인 초기 군집 중심을 선택합니다.
초기 공분산 행렬을 요소 (
j,j)가X(:,j)의 분산인 대각 행렬로 설정합니다.초기 혼합 비율을 균일하게 처리합니다.
각 관측값을 해당 레이블에 연결하여 가우스 혼합 모델을 피팅합니다.
y = ones(size(X,1),1); y(strcmp(species,'setosa')) = 2; y(strcmp(species,'virginica')) = 3; GMModel2 = fitgmdist(X,3,'Start',y);
초기 평균, 공분산 행렬 및 혼합 비율을 명시적으로 지정하여 가우스 혼합 모델을 피팅합니다.
Mu = [1 1; 2 2; 3 3]; Sigma(:,:,1) = [1 1; 1 2]; Sigma(:,:,2) = 2*[1 1; 1 2]; Sigma(:,:,3) = 3*[1 1; 1 2]; PComponents = [1/2,1/4,1/4]; S = struct('mu',Mu,'Sigma',Sigma,'ComponentProportion',PComponents); GMModel3 = fitgmdist(X,3,'Start',S);
gscatter를 사용하여 붓꽃 종 간을 구분하는 산점도 도식을 플로팅합니다. 각 모델에 대해 피팅된 가우스 혼합 모델 등고선을 플로팅합니다.
figure subplot(2,2,1) h = gscatter(X(:,1),X(:,2),species,[],'o',4); haxis = gca; xlim = haxis.XLim; ylim = haxis.YLim; d = (max([xlim ylim])-min([xlim ylim]))/1000; [X1Grid,X2Grid] = meshgrid(xlim(1):d:xlim(2),ylim(1):d:ylim(2)); hold on contour(X1Grid,X2Grid,reshape(pdf(GMModel1,[X1Grid(:) X2Grid(:)]),... size(X1Grid,1),size(X1Grid,2)),20) uistack(h,'top') title('{\bf Random Initial Values}'); xlabel('Sepal length'); ylabel('Sepal width'); legend off; hold off subplot(2,2,2) h = gscatter(X(:,1),X(:,2),species,[],'o',4); hold on contour(X1Grid,X2Grid,reshape(pdf(GMModel2,[X1Grid(:) X2Grid(:)]),... size(X1Grid,1),size(X1Grid,2)),20) uistack(h,'top') title('{\bf Initial Values from Labels}'); xlabel('Sepal length'); ylabel('Sepal width'); legend off hold off subplot(2,2,3) h = gscatter(X(:,1),X(:,2),species,[],'o',4); hold on contour(X1Grid,X2Grid,reshape(pdf(GMModel3,[X1Grid(:) X2Grid(:)]),... size(X1Grid,1),size(X1Grid,2)),20) uistack(h,'top') title('{\bf Initial Values from the Structure}'); xlabel('Sepal length'); ylabel('Sepal width'); legend('Location',[0.7,0.25,0.1,0.1]); hold off
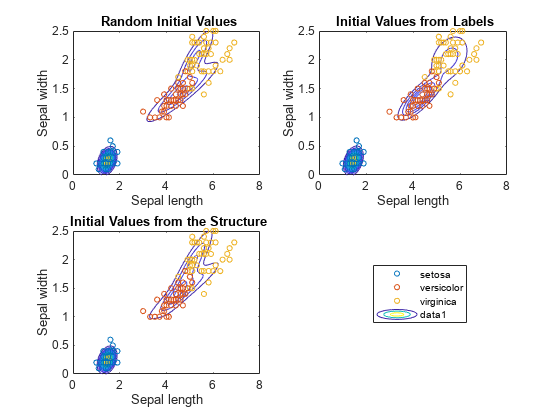
등고선을 보면 GMModel2는 약간의 삼봉 분포를 나타내는 것처럼 보이고, 나머지 모델은 이봉 분포를 나타냅니다.
추정된 성분 평균을 표시합니다.
table(GMModel1.mu,GMModel2.mu,GMModel3.mu,'VariableNames',... {'Model1','Model2','Model3'})
ans=3×3 table
Model1 Model2 Model3
_________________ ________________ ________________
5.2115 2.0119 4.2857 1.3339 1.4604 0.2429
1.461 0.24423 1.462 0.246 4.7509 1.4629
4.6829 1.4429 5.5507 2.0316 5.0158 1.8592
GMModel2가 붓꽃 종 간을 가장 잘 구분하는 것처럼 보입니다.
입력 인수
가우스 혼합 모델을 피팅할 데이터로, 숫자형 행렬로 지정됩니다.
X의 행은 관측값에 대응되고, X의 열은 변수에 대응됩니다. 관측값의 개수는 변수의 개수와 성분의 개수보다 커야 합니다.
NaN은 누락값을 나타냅니다. 소프트웨어는 피팅하기 전에 NaN을 하나 이상 포함하는 X의 행을 제거하여 실질적인 표본 크기를 줄입니다.
데이터형: single | double
가우스 혼합 모델을 피팅할 때 사용할 성분 개수로, 양의 정수로 지정됩니다. 예를 들어, k = 3을 지정하면 소프트웨어는 각각 세 개의 고유한 평균, 공분산 행렬 및 성분 비율을 갖는 가우스 혼합 모델을 데이터(X)에 피팅합니다.
데이터형: single | double
이름-값 인수
선택적 인수 쌍을 Name1=Value1,...,NameN=ValueN으로 지정합니다. 여기서 Name은 인수 이름이고 Value는 대응값입니다. 이름-값 인수는 다른 인수 뒤에 와야 하지만, 인수 쌍의 순서는 상관없습니다.
R2021a 이전 릴리스에서는 쉼표를 사용하여 각 이름과 값을 구분하고 Name을 따옴표로 묶으십시오.
예: 'RegularizationValue',0.1,'CovarianceType','diagonal'은 정규화 모수 값으로 0.1을 지정하고 대각 공분산 행렬을 피팅하도록 지정합니다.
데이터에 피팅할 공분산 행렬 유형으로, 'CovarianceType'과 함께 'diagonal' 또는 'full'이 쉼표로 구분되어 지정됩니다.
'diagonal'을 설정하면 소프트웨어는 대각 공분산 행렬을 피팅합니다. 이 경우, 소프트웨어는 k*d개의 공분산 모수를 추정합니다. 여기서 d는 X에 포함된 열의 개수입니다(즉, d = size(X,2)).
그렇지 않은 경우, 소프트웨어는 완전 공분산 행렬을 피팅합니다. 이 경우, 소프트웨어는 k*d*(d+1)/2개의 공분산 모수를 추정합니다.
예: 'CovarianceType','diagonal'
반복 EM 알고리즘 최적화 옵션으로, 'Options'와 함께 statset options 구조체가 쉼표로 구분되어 지정됩니다.
다음 표에는 사용 가능한 이름-값 쌍의 인수가 설명되어 있습니다.
| 이름 | 값 |
|---|---|
'Display' |
|
'MaxIter' | 허용되는 최대 반복 횟수를 나타내는 양의 정수입니다. 디폴트 값은 100입니다. |
'TolFun' | 로그 가능도 함수 값에 대한 종료 허용오차를 나타내는 양의 스칼라입니다. 디폴트 값은 1e-6입니다. |
예: 'Options',statset('Display','final','MaxIter',1500,'TolFun',1e-5)
사후 확률에 대한 허용오차로, ProbabilityTolerance와 [0,1e-6] 범위의 음이 아닌 스칼라 값이 쉼표로 구분되어 지정됩니다.
각 반복마다 사후 확률을 추정한 후 fitgmdist는 허용오차 값보다 크지 않은 사후 확률을 0으로 설정합니다. 0이 아닌 허용오차를 사용하면 fitgmdist의 속도를 높일 수 있습니다.
예: 'ProbabilityTolerance',0.0000025
데이터형: single | double
정규화 모수 값으로, 'RegularizationValue'와 함께 음이 아닌 스칼라가 쉼표로 구분되어 지정됩니다.
RegularizationValue를 작은 양의 스칼라로 설정하여 추정된 공분산 행렬이 양의 정부호 행렬이 되도록 합니다.
예: 'RegularizationValue',0.01
데이터형: single | double
새 초기값 세트를 사용하여 EM 알고리즘을 반복하는 횟수로, 'Replicates'와 함께 양의 정수가 쉼표로 구분되어 지정됩니다.
Replicates가 1보다 큰 경우 다음과 같습니다.
이름-값 쌍의 인수
Start는plus(디폴트 값) 또는randSample이어야 합니다.GMModel은 최대 로그 가능도를 갖는 피팅입니다.
예: 'Replicates',10
데이터형: single | double
모든 공분산 행렬이 동일한지 여부를 나타내는 플래그(즉, 합동 추정값을 피팅함)로, 'SharedCovariance'와 함께 논리값 false 또는 true가 쉼표로 구분되어 지정됩니다.
SharedCovariance가 true이면 k개 공분산 행렬이 모두 같고, 공분산 모수 개수가 k배만큼 축소됩니다.
초기값 설정 방법으로, 'Start'와 함께 'randSample', 'plus', 정수로 구성된 벡터 또는 구조체형 배열이 쉼표로 구분되어 지정됩니다.
Start의 값에 따라 평균, 공분산, 혼합 비율 등의 각 가우스 성분 모수에 대해 최적화 루틴에 필요한 초기값이 결정됩니다. 다음 표에는 사용 가능한 옵션이 요약되어 있습니다.
| 값 | 설명 |
|---|---|
'randSample' | 소프트웨어가 X에서 임의로 k개 관측값을 초기 성분 평균으로 선택합니다. 혼합 비율은 균일합니다. 모든 성분에 대한 초기 공분산 행렬은 대각선상의 요소 j가 X(:,j)의 분산인 대각 행렬입니다. |
'plus' | 소프트웨어가 k-평균++ 알고리즘을 사용하여 X에서 k개 관측값을 선택합니다. 초기 혼합 비율은 균일합니다. 모든 성분에 대한 초기 공분산 행렬은 대각선상의 요소 j가 X(:,j)의 분산인 대각 행렬입니다. |
| 정수로 구성된 벡터 | 각 점의 성분 인덱스에 대한 초기 추측값을 포함하는 길이가 n(관측값 개수)인 벡터입니다. 즉, 각 요소는 성분에 대응되는, 1에서 k 사이의 정수입니다. 소프트웨어는 동일한 성분에 대응되는 모든 관측값을 수집하고, 각 성분에 대해 평균, 공분산 및 혼합 비율을 계산하며, 초기값을 이러한 통계량으로 설정합니다. |
| 구조체형 배열 |
|
예: 'Start',ones(n,1)
데이터형: single | double | char | string | struct
출력 인수
피팅된 가우스 혼합 모델로, gmdistribution 모델로 반환됩니다.
점 표기법을 사용하여 GMModel의 속성에 액세스할 수 있습니다. 예를 들어, GMModel.AIC를 입력하면 AIC를 표시할 수 있습니다.
팁
fitgmdist 함수는 다음과 같을 수 있습니다.
하나 이상의 성분에 조건이 나쁜 공분산 행렬 또는 특이 공분산 행렬이 있는 경우 해로 수렴할 수 있습니다.
다음과 같은 문제로 인해 조건이 나쁜 공분산 행렬이 생성될 수 있습니다.
데이터의 차원 수가 비교적 높고 관측값 개수가 충분하지 않습니다.
데이터의 일부 예측 변수 간에 높은 상관관계가 있습니다.
일부 또는 모든 특징이 이산적입니다.
너무 많은 성분에 데이터를 피팅하려고 했습니다.
일반적으로 다음 예방책 중 하나를 사용하여 조건이 나쁜 공분산 행렬이 생성되는 것을 피할 수 있습니다.
데이터를 전처리하여 상관관계가 있는 특징을 제거합니다.
'SharedCovariance'를true로 설정하여 모든 성분에 동일한 공분산 행렬을 사용합니다.'CovarianceType'를'diagonal'로 설정합니다.'RegularizationValue'를 사용하여 매우 작은 양수를 모든 공분산 행렬의 대각선에 더합니다.다른 초기값 세트를 사용해 봅니다.
하나 이상의 성분에 조건이 나쁜 공분산 행렬이 있는 경우 중간 과정을 건너뛸 수 있습니다. 다른 초기값 세트를 사용하여 데이터 또는 모델을 변경하지 않고 이 문제를 방지할 수 있습니다.
알고리즘
소프트웨어는 반복 기대값 최대화(EM) 알고리즘을 사용하여 가우스 혼합 모델 가능도를 최적화합니다.
fitgmdist는 반복 기대값 최대화 알고리즘을 사용하여 가우스 혼합 모델을 데이터에 피팅합니다. EM 알고리즘은 성분 평균, 공분산 행렬 및 혼합 비율에 대한 초기값을 사용하여 다음 단계를 수행합니다.
각 관측값에 대해 이 알고리즘은 각 관측값이 특정 성분에 속할 사후 확률을 계산합니다. 결과를 n×k 행렬로 간주할 수 있습니다. 여기서 행렬의 요소 (i,j)는 관측값 i가 성분 j에 속할 사후 확률을 포함합니다. 이는 EM 알고리즘의 E 단계입니다.
성분 소속 사후 확률을 가중치로 사용하는 이 알고리즘은 최대가능도를 적용하여 성분 평균, 공분산 행렬 및 혼합 비율을 추정합니다. 이는 EM 알고리즘의 M 단계입니다.
이 알고리즘은 수렴할 때까지 이러한 단계를 반복합니다. 가능도 곡면은 복잡하며 알고리즘이 국소 최적해로 수렴할 수 있습니다. 또한, 결과로 생성된 국소 최적해는 초기 조건에 따라 달라질 수 있습니다. fitgmdist에는 관측값에 대한 임의 성분 할당, k-평균++ 알고리즘 등 초기 조건을 선택하는 데 사용할 수 있는 여러 가지 옵션이 있습니다.
k-평균++ 알고리즘은 발견적 방식을 사용하여 k-평균 군집화에 사용할 중심 시드값을 구합니다. fitgmdist는 k-평균++ 알고리즘을 사용하여 피팅된 가우스 혼합 모델에 사용할 초기 모수 값을 선택함으로써 EM 알고리즘을 초기화하는 데 이와 동일한 원리를 적용할 수 있습니다.
k-평균++ 알고리즘은 군집 개수가 k라고 가정하고 다음과 같이 초기 모수 값을 선택합니다.
성분 혼합 확률이 균등 확률 (여기서 i = 1, ..., k임)이 되도록 선택합니다.
공분산 행렬이 대각 행렬이며 동일한 행렬(여기서 이고 임)이 되도록 선택합니다.
X의 모든 데이터 점에서 대해 똑같이 첫 번째 초기 성분 중심 μ1을 선택합니다.
다음을 수행하여 중심 j를 선택합니다.
각 관측값에서 각 중심까지의 마할라노비스 거리를 계산하고 각 관측값을 이에 가장 가까운 중심에 할당합니다.
m = 1,...,n 및 p = 1,...,j – 1에 대해 다음 확률로 X에서 임의로 중심 j를 선택합니다.
여기서 는 관측값 m과 μp 사이의 거리이고, Mp는 중심 μp에 가장 가까운 모든 관측값의 집합이며, xm은 Mp에 속합니다.
즉, 이미 선택한 가장 가까운 중심과의 거리에 비례하는 확률로 각 후속 중심을 선택합니다.
k개 중심이 선택될 때까지 4번째 단계를 반복합니다.
참고 문헌
[1] McLachlan, G., and D. Peel. Finite Mixture Models. Hoboken, NJ: John Wiley & Sons, Inc., 2000.
버전 내역
R2014a에 개발됨
MATLAB Command
You clicked a link that corresponds to this MATLAB command:
Run the command by entering it in the MATLAB Command Window. Web browsers do not support MATLAB commands.
웹사이트 선택
번역된 콘텐츠를 보고 지역별 이벤트와 혜택을 살펴보려면 웹사이트를 선택하십시오. 현재 계신 지역에 따라 다음 웹사이트를 권장합니다:
또한 다음 목록에서 웹사이트를 선택하실 수도 있습니다.
사이트 성능 최적화 방법
최고의 사이트 성능을 위해 중국 사이트(중국어 또는 영어)를 선택하십시오. 현재 계신 지역에서는 다른 국가의 MathWorks 사이트 방문이 최적화되지 않았습니다.
미주
- América Latina (Español)
- Canada (English)
- United States (English)
유럽
- Belgium (English)
- Denmark (English)
- Deutschland (Deutsch)
- España (Español)
- Finland (English)
- France (Français)
- Ireland (English)
- Italia (Italiano)
- Luxembourg (English)
- Netherlands (English)
- Norway (English)
- Österreich (Deutsch)
- Portugal (English)
- Sweden (English)
- Switzerland
- United Kingdom (English)