이 페이지는 기계 번역되었습니다.
번역 품질에 대한 1분 설문 조사에 참여해 주세요.
보조 병리 진단을 위한 딥러닝 모델 개발
작성자: Ong Kok Haur, Laurent Gole, Huo Xinmi, Li Longjie, Lu Haoda, Yuen Cheng Xiang, Aisha Peng 및 Yu Weimiao, Bioinformatics Institute
전립선암은 남성들 사이에서 두 번째로 많이 발병하는 질병이며 일반적으로 조직 표본을 검사하여 진단합니다. 이 과정은 전통적으로 전문 병리학자가 현미경을 사용하여 수행하는데, 노동 집약적이고 시간이 많이 소요됩니다. 더욱이 이런 검사를 수행할 수 있는 병리학자 등 의료 전문가의 수는 많은 국가에서 제한적이며, 특히 임상 작업량이 많은 경우에는 더욱 그렇습니다. 이로 인해 분석해야 할 표본이 적체되고 치료 개시가 지연될 수 있습니다.
수동으로 표본을 분석하는 데 한계가 있는 것도 이유 중 하나지만, 전립선암 및 기타 형태의 암에 대한 병리학적 진단을 견인하기 위해 AI와 딥러닝을 사용하는 연구가 급속히 확대되었습니다. 그러나 딥러닝 모델을 개발, 최적화, 검증하고 임상에 적용하기 위해서는 몇 가지 기술적 장벽을 극복해야 합니다. 예를 들어, 디지털 병리학 영상의 약 15%는 초점, 채도, 아티팩트와 관련된 품질 문제가 있는 것으로 추정됩니다. 게다가 영상 품질은 육안으로 정량적 평가가 불가능하며, 최근 활용되는 WSI(전체 슬라이드 영상) 스캐너는 매우 큰 대규모 데이터셋을 생성하기 때문에 85,000×40,000픽셀 이상의 고해상도 영상으로 인해 영상 처리가 복잡해질 수 있습니다. 또한 수동 병리 진단과 마찬가지로 영상에 주석을 다는 과정은 숙련된 병리학자에게도 상당한 시간이 필요합니다. 이 과정으로 인해 정확도가 높은 딥러닝 모델의 훈련에 필요한 양질의 레이블 지정 영상 데이터베이스를 구축하기가 어렵습니다.
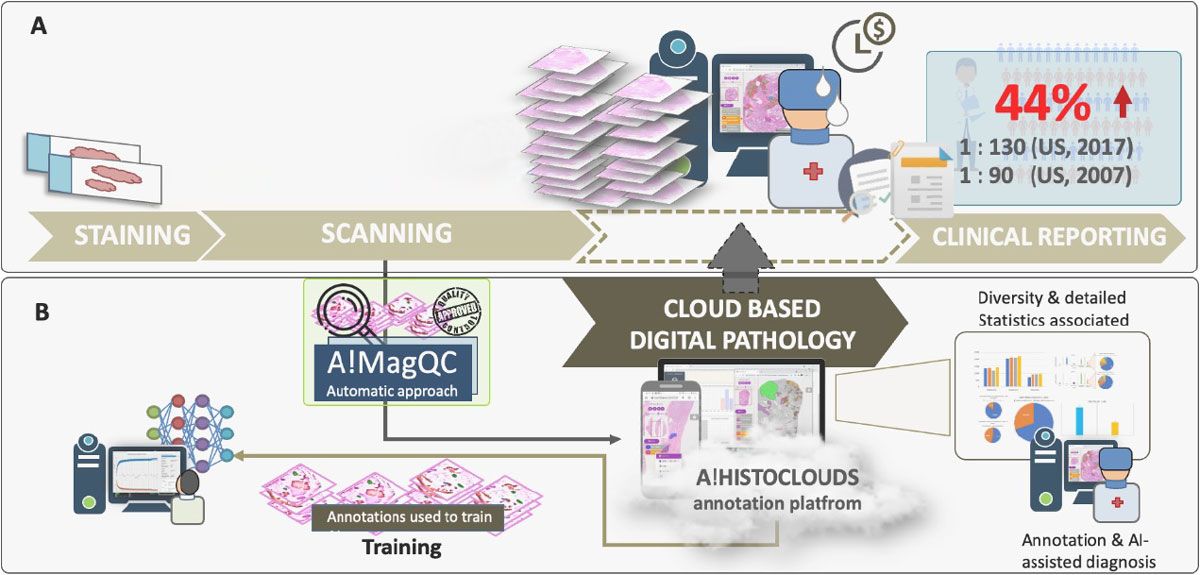
A*STAR 산하의 BII(Bioinformatics Institute)의 CDPL(Computational Digital Pathology Lab)은 딥러닝 보조 병리 진단과 관련된 여러 문제를 해결하는 동시에 영상 레이블 지정 및 임상 진단에 대한 병리학자의 부담을 줄여 주는 클라우드 기반 자동화 플랫폼을 개발했습니다. (그림 1) 이 플랫폼에는 Deep Learning Toolbox™ 및 Image Processing Toolbox™를 사용하여 MATLAB®에서 개발된 완전 자동 영상 품질 평가 툴인 A!MagQC가 포함되어 있습니다. 이 플랫폼에는 글리슨 패턴을 식별하도록 훈련된 딥러닝 분류 모델도 포함되어 있습니다. 국내외 병리학자를 대상으로 실시한 실험에서 이 플랫폼은 수동 주석 및 전통적인 현미경 검사에 비해 영상 레이블링 시간을 60% 단축시켜, 병리학자가 전통적인 현미경 검사와 동일한 정확도를 유지하면서도 영상을 43% 더 빠르게 분석할 수 있도록 도왔습니다.
영상 품질 평가
디지털 병리에서 영상 품질 문제는 크게 조직 표본 준비 문제와 스캐닝 문제의 두 범주로 나눌 수 있습니다. (그림 2) 조직 파열, 주름, 기포, 과염색 및 과소염색이 첫 번째 범주에 해당하며, 이러한 문제가 발견되어 진단에 영향을 미치는 경우 새로운 표본을 준비해야 합니다. 반면에 영상 대비, 채도, 초점 문제가 검출되면 기존 표본을 다시 스캔하기만 하면 되므로 재절단할 필요가 없습니다.
병리학자가 하는 분석이든 딥러닝 모델을 통한 분석이든, 흔히 발생하는 이런 문제들은 부정적인 영향을 가질 수 있습니다. 이에 따라 A*STAR의 BII CDPL팀은 A!MagQC에서 영상 품질에 영향을 미치는 주요 요소를 자동으로 감지하는 영상 처리 알고리즘을 개발했습니다. 이 팀은 특화된 툴박스 때문에 MATLAB을 선택했습니다. 예를 들어, 영상이 메모리로 불러오기에는 너무 큰 경우 Image Processing Toolbox의 blockproc 함수를 사용하면 각 영상을 지정된 크기의 블록으로 나누고, 한 번에 한 블록씩 처리한 다음 결과를 조립하여 출력 영상을 만들 수 있습니다.
또한 팀은 MATLAB 툴을 사용하여 A!MagQC 사용자 인터페이스를 구축하고 배포를 위해 MATLAB 코드를 독립형 A!MagQC 실행 파일로 컴파일했습니다.
연구팀은 개발된 QC 솔루션을 사용해 WSI의 색상, 밝기, 대비의 차이를 파악하기 위해 영상 품질을 정량화했습니다. 이 연습을 통해 이후 훈련된 딥러닝 모델이 오늘날 사용되는 다양한 스캐너에 대해 정확한 진단 결과를 생성할 수 있음이 확인되었습니다.
모델 훈련 및 테스트
병리학자들은 표본을 분석할 때 전립선암에 특화된 글리슨 분류법을 적용하여 모양에 따라 점수를 부여합니다. 정상 또는 양성 조직 외에 간질 (결합 조직) 또는 1~5점까지의 글리슨 점수가 부여된 조직이 포함될 수 있는데, 여기서 5점은 가장 비정상적임을 뜻합니다. (그림 3) 연구진이 조직 표본을 분류하기 위한 AI 진단 모델 훈련을 시작하기에 앞서, 이러한 범주로 레이블이 지정된 영상 패치의 데이터셋을 조립해야 했습니다. 이 작업은 A!MagQC를 사용하여 품질이 검사된 영상을 처리하는 A!HistoClouds를 사용하는 병리학자의 도움으로 완료되었습니다. 연구팀은 레이블이 지정된 영상 패치의 기본 세트를 얻은 후, 개별 영상을 수직 또는 수평으로 반사시키고 무작위 또는 지정된 각도만큼 회전시켜서 데이터 증강을 수행하여 훈련 세트를 확장했습니다.
팀은 Deep Learning Toolbox를 사용하여 MATLAB에서 작업하여 ResNet-50, VGG-16, NasNet-Mobile의 사전 학습된 신경망을 사용하여 딥러닝 모델 구조를 만들고 일반 분류 계층을 가중 분류 계층으로 대체했습니다. (그림 4). 또한 우리는 딥러닝 모델 훈련에서 단일 GPU에서 여러 GPU로 확장하기 위해 multi-gpu 옵션을 사용했습니다.
모델은 반복적인 과정을 통해 훈련되고 적용됩니다. 수작업으로 레이블 지정된 영상에 대한 초기 훈련이 이루어지는 첫 번째 단계 이후에는 훈련된 모델을 통해 생성된 예측 결과를 병리학자가 검토 및 수정하는 반자동식의 두 번째 단계로 넘어갑니다. (그림 5) 두 번째 단계는 의료 전문가가 임상 진단을 돕기 위해 모델을 활용할 준비가 될 때까지 반복됩니다. (a) 단계에서는 초급 및 고급 병리학자 모두가 수동으로 주석을 달도록 요구됩니다. 주석은 A!HistoClouds를 사용하여 작성되며, 이를 패치로 추출하여 딥러닝 모델을 훈련하는 데 사용합니다. 이 모델은 병리학자를 돕기 위해 예측된 ROI(관심 영역)를 출력하는데, 이를 반자동 주석이라고 합니다. (b) 단계에서는 모델이 점진적 학습을 거칩니다. 즉, AI가 예측한 ROI를 병리학자가 검토하여 수정하고, ROI를 패치로 추출하고, 모델은 이 새로운 데이터로부터 학습합니다. (b) 단계는 모델 성능이 수렴할 때까지 반복되며, (c) 단계에서는 병리학자의 의사결정을 향상시키는 완전 자동 주석/완전 자동 진단을 달성하는 데 모델이 배포됩니다.
다음 단계
CDPL은 그 이후 딥러닝 지원 병리학 진단 플랫폼을 글로벌 클라우드 플랫폼에 구축하여 다양한 국가에서 근무하는 병리학자 팀에게 쉬운 접근성을 제공했습니다. A*STAR의 BII는 현재 다양한 조직 두께, 염색 메커니즘, 영상 스캐너를 포함한 추가적인 임상 시나리오에 대한 딥러닝 모델의 검증 및 최적화 작업을 진행하고 있습니다. 마지막으로 BII는 동일한 영상 품질 평가 및 딥러닝 워크플로를 전립선암뿐 아니라 다른 유형의 암에도 확장할 수 있는 기회를 모색하고 있습니다.
BII의 CDPL은 또한 AGGC 2022(Automated Gleason Grading Challenge 2022)를 조직했는데, 이는 2022 International Conference on Medical Image Computing and Computer Assisted Intervention에서 채택되었습니다. AGGC 2022는 전립선암의 글리슨 분류, 디지털 병리 활용, 딥러닝 접근법과 관련된 도전 과제를 해결하는 데 초점을 맞추고 있습니다. 이 챌린지의 목표는 다양한 디지털 스캐너에서 확보한 H&E 염색 전립선 조직병리학 영상에 대해 정확도가 우수한 자동 알고리즘을 개발하는 것입니다. 특히 이 챌린지는 디지털 병리 분야에서 최초로 영상 스캐닝 과정에 따른 색상 변화를 조사했습니다.
챌린지는 종료되었지만 전체 데이터셋은 현재 지속적인 연구에 활용할 수 있습니다.
감사의 글
A*STAR의 BII는 NUH(National University Hospital)의 동료들, 특히 Tan Soo Yong 교수와 Susan Hue Swee Shan 박사, Lau Kah Weng 박사, Tan Char Loo 박사 등의 파트너십과 협력에 감사드리고 싶습니다. NUH는 이 연구에 사용된 데이터와 표본의 출처로 정당하게 인정받았습니다. 팀은 다른 임상 및 산업 파트너로부터 받은 지원에 감사하고 있습니다.
2024년 기고