불규칙적으로 샘플링된 시계열 데이터로 잠재 ODE 신경망 훈련시키기
이 예제에서는 불규칙적인 시간 간격으로 샘플링된 시계열 데이터를 사용하여 잠재(latent) 상미분 방정식(ODE) 오토인코더를 훈련시키는 방법을 보여줍니다.
시계열 데이터를 위한 대다수 딥러닝 모델(예: 순환 신경망)은 그 훈련을 위해 규칙적으로 샘플링된 시계열 데이터를 필요로 합니다. 즉, 시퀀스의 요소가 고정 너비 시간 간격에 대응해야 합니다.
불규칙적으로 샘플링된 시계열 데이터의 동특성을 학습하기 위해 잠재 ODE 모델 [1, 2]를 사용할 수 있습니다. 잠재 ODE 모델은 시계열 데이터의 동특성을 학습하는 변분 오토인코더(VAE)[3]입니다. 오토인코더는 입력값을 복제하도록 훈련시킨 모델 유형으로, 입력값을 잠재 공간으로 변환하고(인코딩 단계) 잠재 표현으로부터 입력값을 복원합니다(디코딩 단계). 오토인코더를 훈련시킬 때 레이블이 지정된 데이터가 필요하지 않습니다.
대다수 오토인코더와 달리, 잠재 ODE 모델은 입력을 정확히 복제하도록 훈련되지 않습니다. 대신 이 모델은 입력 데이터의 동특성을 학습하며, 사용자는 모델이 대응되는 값을 예측할 일련의 목표 타임스탬프를 지정할 수 있습니다.
다음 도식은 모델의 구조를 보여줍니다.

잠재 ODE 모델을 훈련시키는 데에는 많은 시간이 소요됩니다. 기본적으로 이 예제에서는 훈련을 건너뛰고, 사전 훈련된 모델을 불러옵니다. 그러지 않고 모델을 훈련시키려면 doTraining 플래그를 true로 설정하십시오.
doTraining = false;
데이터 불러오기
Irregular Sine Waves 데이터 세트를 불러옵니다. 이 데이터 세트에는 다양한 주파수와 오프셋, 잡음으로 생성된 합성 사인파 1000개가 들어 있습니다. 각 시퀀스에는 불규칙하게 샘플링된 동일한 타임스탬프가 사용됩니다.
load irregularSineWaves루프를 사용해 채널을 순회하여 첫 번째 시퀀스를 플롯으로 시각화합니다. 타임스탬프를 강조 표시하는 세로선을 플로팅합니다. Irregular Sine Waves 데이터 세트는 채널을 하나만 포함하기 때문에 다음 코드는 단일 플롯만 표시합니다.
numChannels = size(values,1); idx = 1; figure t = tiledlayout(numChannels,1); title(t,"Observation " + idx) for i = 1:numChannels nexttile plot(tspan,squeeze(values(i,idx,:)),Marker="."); xlabel("t") ylabel("Value") xline(tspan,":") end

훈련을 위해 데이터 준비하기
이 예제에 지원 파일로 첨부된 trainingPartitions 함수를 사용하여 훈련 데이터와 테스트 테이터를 분할합니다. 이 함수에 액세스하려면 예제를 라이브 스크립트로 여십시오. 데이터의 80%는 훈련용, 나머지 20%는 테스트용으로 사용합니다.
numObservations = size(values,2); [idxTrain,idxTest] = trainingPartitions(numObservations,[0.8 0.2]); sequencesTrain = values(:,idxTrain,:); sequencesTest = values(:,idxTest,:);
훈련 데이터와 테스트 데이터를 출력하는 데이터저장소를 만듭니다.
dsTrain = arrayDatastore(sequencesTrain,IterationDimension=2); dsTest = arrayDatastore(sequencesTest,IterationDimension=2);
모델의 학습 가능한 파라미터 초기화하기
이 예제에서는 VAE를 훈련시킵니다. 여기서 인코더는 ODE-RNN[2]이라고 하는 순환 신경망(RNN)을 사용하며 디코더는 신경망 ODE입니다. 인코더는 시퀀스를 고정 길이 잠재 표현으로 매핑합니다. 이 잠재 표현은 가우스 분포를 파라미터화합니다. 모델은 인코딩된 이러한 파라미터를 사용하여 가우스 분포에서 샘플링한 다음, 샘플링된 데이터를 디코더에 전달합니다.
다음 도식은 모델의 구조를 보여줍니다.
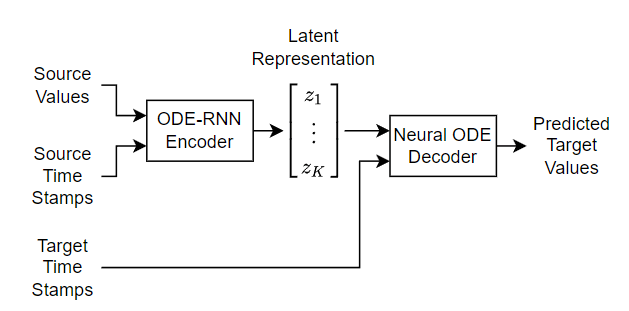
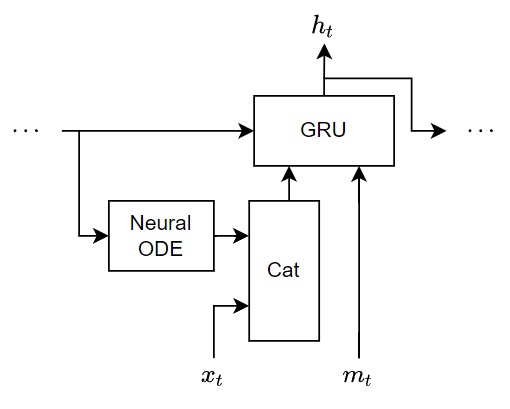
인코더는 ODE-RNN입니다. 이 인코더는 인코더에 대한 마지막 입력값이 디코더에 대한 첫 번째 출력값이 되도록 입력 시퀀스의 순서를 뒤집습니다. ODE-RNN은 순서를 뒤집은 데이터의 각 시간 스텝을 마스크 처리된 게이트 순환 유닛(GRU)과 ODE 솔버를 사용해 읽어 들이면서 잠재 표현을 업데이트합니다. ODE-RNN은 입력 데이터와 GRU 연산의 은닉 상태를 결합하고, 신경망 ODE(Neural ODE)를 기반으로 한 GRU 연산의 출력값을 다음 시간 스텝으로 진전시킵니다. GRU 연산은 마스크로 지정된 시간 스텝에서만 업데이트된 상태를 사용합니다.
다음 도식은 인코더의 구조를 보여줍니다.

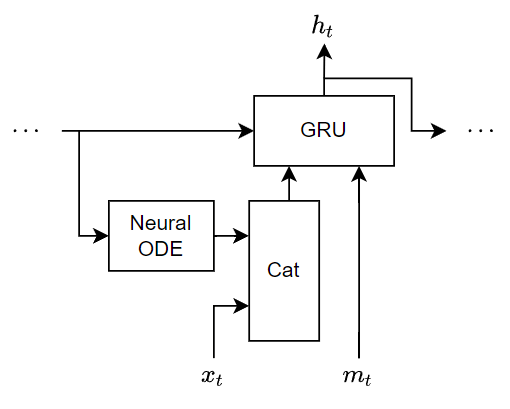
다음 도식은 데이터의 시간 스텝을 처리할 때의 ODE-RNN 구조를 보여줍니다. 이 도식에서 는 시간 스텝의 값을 나타내고, 는 마스크 값을 나타내며, 는 GRU 연산의 은닉 상태 출력값입니다.
디코더는 잠재 표현을 취하고, 지정된 목표 타임스탬프에 맞춰 입력 시퀀스를 복원합니다. 디코더는 잠재 표현과 목표 타임스탬프를 신경망 ODE에 전달하고 신경망 ODE 출력값을 잠재 표현과 결합한 다음, 결합된 데이터를 완전 연결 연산에 전달하고, 입력 시퀀스와 일치하도록 출력값의 형태를 변경합니다.
다음 도식은 디코더의 구조를 보여줍니다.

모델을 훈련시키기 위해, 잠재 ODE를 위한 학습 가능한 파라미터가 포함된 구조를 만듭니다. 이 예제에 지원 파일로 첨부된 latentODEParameters 함수를 사용하여 모델의 학습 가능한 파라미터를 초기화합니다. 이 함수에 액세스하려면 예제를 라이브 스크립트로 여십시오. 이 함수는 좁은 정규분포(평균이 0이고 표준편차가 0.01인 가우스 분포)에서 샘플링하여, 완전 연결 연산과 GRU 연산을 위한 학습 가능한 파라미터를 초기화합니다.
모델 하이퍼파라미터를 다음과 같이 지정합니다.
인코더 ODE 크기: 105
인코더 RNN 크기: 40
디코더 ODE 크기: 110
잠재 크기: 32
encoderODEHiddenSize = 100; encoderRNNHiddenSize = 40; decoderODEHiddenSize = 100; latentSize = 10; inputSize = numChannels; parameters = latentODEParameters(inputSize,encoderODEHiddenSize,encoderRNNHiddenSize,latentSize,decoderODEHiddenSize);
모델 함수 정의하기
딥러닝 모델에 사용할 모델 함수를 정의합니다.
모델 함수
model 함수(이 예제의 모델 손실 함수 섹션에 정의되어 있음)는 모델의 학습 가능한 파라미터, 소스 타임스탬프 및 그에 대응되는 시퀀스 값과 마스크, 목표 타임스탬프를 입력값으로 받습니다. 이 함수는 목표 타임스탬프에 대응되는 예측값을 반환합니다.
인코더 함수
encoder 함수(이 예제의 인코더 함수 섹션에 정의되어 있음)는 인코더의 학습 가능한 파라미터, 소스 타임스탬프 및 그에 대응되는 시퀀스 값과 마스크를 입력값으로 받습니다. 이 함수는 잠재 표현을 반환합니다.
디코더 함수
decoder 함수(이 예제의 디코더 함수 섹션에 정의되어 있음)는 디코더의 학습 가능한 파라미터, 목표 타임스탬프 및 잠재 표현을 입력값으로 받습니다. 이 함수는 목표 타임스탬프에 대응되는 예측값을 반환합니다.
모델 손실 함수 정의하기
modelLoss 함수(이 예제의 모델 손실 함수 섹션에 정의되어 있음)는 모델의 학습 가능한 파라미터, 타임스탬프 및 그에 대응되는 시퀀스 값과 마스크를 입력값으로 받습니다. 이 함수는 모델 손실과 학습 가능한 파라미터에 대한 손실 기울기를 반환합니다. 모델 손실 함수는 입력 관측값 수과 샘플 수에 대해 정규화된 손실을 사용합니다.
훈련 옵션 지정하기
다음과 같이 훈련 옵션을 지정합니다.
크기가 50인 미니 배치로 Epoch 200회만큼 훈련시킵니다.
초기 학습률 0.0025로 훈련시킵니다.
학습률이 0.00025에 도달할 때까지 각 반복마다 0.999의 비율로 학습률을 기하급수적으로 감쇠시킵니다.
CPU를 사용하여 훈련시킵니다. 경우에 따라 신경망 ODE 모델의 훈련 속도가 GPU에서보다 CPU에서 더 빠를 수 있습니다.
numEpochs = 200;
miniBatchSize = 50;
initialLearnRate = 2.5e-3;
minLearnRate = 2.5e-4;
decayRate = 0.999;
executionEnvironment = "cpu";모델 훈련시키기
손실 함수 modelLoss를 사용하여 사용자 지정 훈련 루프에서 모델을 훈련시킵니다.
데이터를 읽도록 minibatchqueue 객체를 구성합니다.
이 예제의 미니 배치 전처리 함수 섹션에 나와 있는
preprocessMiniBatch함수를 사용하여 미니 배치를 전처리합니다. 이 함수는 시간 스텝을 무작위로 제거한 시퀀스로 구성된 미니 배치와 그에 대응되는 마스크 및 목표값 시퀀스를 출력합니다.미니 배치 출력 형식을
"CBT"(채널, 배치, 시간)로 지정합니다.미니 배치 출력의 하드웨어 환경을 지정합니다.
numOutputs = 3; mbqTrain = minibatchqueue(dsTrain,numOutputs,... MiniBatchSize=miniBatchSize,... MiniBatchFcn=@preprocessMiniBatch,... MiniBatchFormat=["CBT" "CBT" "CBT"], ... OutputEnvironment=[executionEnvironment executionEnvironment executionEnvironment]);
학습률을 초기화합니다.
learnRate = initialLearnRate;
Adam 솔버에 대한 파라미터를 초기화합니다.
trailingAvg = []; trailingAvgSq = [];
훈련 진행 상황 모니터의 진행률 표시줄을 업데이트하기 위해 총 훈련 반복 횟수를 계산합니다.
numObservationsTrain = size(sequencesTrain,2); numIterationsPerEpoch = ceil(numObservationsTrain/miniBatchSize); numIterations = numIterationsPerEpoch * numEpochs;
사용자 지정 훈련 루프에서 모델을 훈련시킵니다. 각 Epoch마다 훈련 데이터를 섞습니다.
훈련 데이터로 구성된 미니 배치를 루프를 사용해 순환합니다. 각 반복에 대해 다음을 수행합니다.
지수 감쇠를 사용하여 학습률을 업데이트합니다.
dlfeval함수와modelLoss함수를 사용하여 모델 손실과 모델 기울기를 계산합니다.adamupdate함수를 사용하여 학습 가능한 파라미터를 업데이트합니다.훈련 진행 상황 모니터에서 훈련 손실을 기록합니다.
if doTraining % Initialize the training progress monitor. monitor = trainingProgressMonitor( ... Metrics="TrainingLoss", ... Info=["LearnRate" "Epoch"]); monitor.XLabel = "Iteration"; % Loop over the epochs. epoch = 0; iteration = 0; while epoch < numEpochs && ~monitor.Stop epoch = epoch + 1; % Shuffle the training data. shuffle(mbqTrain); % Loop over the training data. while hasdata(mbqTrain) && ~monitor.Stop iteration = iteration + 1; % Update the learning rate. learnRate = max(decayRate*learnRate,minLearnRate); % Read a mini-batch of data. [X,mask,T] = next(mbqTrain); % Calculate the model loss and gradients. [loss,gradients] = dlfeval(@modelLoss,parameters,tspan,X,mask,T); % Update the learnable parameters. [parameters,trailingAvg,trailingAvgSq] = adamupdate(parameters,gradients, ... trailingAvg,trailingAvgSq,iteration,learnRate); % Update the training progress monitor. recordMetrics(monitor,iteration,TrainingLoss=loss); updateInfo(monitor,LearnRate=learnRate,Epoch=(epoch+" of "+numEpochs)); monitor.Progress = 100*(iteration/numIterations); end end % Save the model. save("irregularSineWavesParameters.mat","parameters","tspan"); else s = load("irregularSineWavesParameters.mat"); parameters = s.parameters; miniBatchSize = s.miniBatchSize; end

모델 테스트하기
테스트 데이터를 사용하여 시간 스텝을 시퀀스에서 무작위로 제거한 미니 배치 대기열을 만들어 모델을 테스트하고, 훈련된 잠재 ODE 모델을 사용하여 제거된 값을 예측합니다.
테스트 데이터를 전처리하는 미니 배치 대기열을 훈련 데이터와 동일한 단계를 사용하여 만듭니다.
numOutputs = 2; mbqTest = minibatchqueue(dsTest,numOutputs,... MiniBatchSize=miniBatchSize, ... MiniBatchFcn=@preprocessMiniBatch, ... MiniBatchFormat=["CBT" "CBT"], ... OutputEnvironment=[executionEnvironment executionEnvironment]);
원래 입력 타임스탬프와 일치하도록 목표 타임스탬프를 지정합니다.
tspanTarget = tspan;
루프를 사용해 미니 배치 대기열을 순회하고 모델에 데이터를 통과시켜 예측을 수행합니다.
YTest = []; while hasdata(mbqTest) [X,mask] = next(mbqTest); Y = model(parameters,tspan,X,tspanTarget,Mask=mask); YTest = cat(2,YTest,Y); end
RMS 오차를 계산합니다.
rmse = sqrt(mean((sequencesTest - YTest).^2,"all"))rmse =
1(C) × 1(B) × 1(T) single dlarray
0.1589
오차를 히스토그램으로 시각화합니다.
err = sequencesTest - YTest; figure err = extractdata(err); histogram(err) xlabel("Error") ylabel("Frequency") title("Test RMSE = " + string(rmse))

새 데이터를 사용하여 예측하기
0과 5 사이의 균일한 간격의 타임스탬프 1000개를 사용하여 테스트 시퀀스를 복원합니다.
테스트 데이터를 포함한 미니 배치 대기열을 만듭니다.
어떠한 시간 스텝도 제거하지 않은 채로 시퀀스 데이터로 구성된 미니 배치를 생성하는 preprocessMiniBatchPrtedictors 함수를 사용하여 데이터를 전처리합니다.
numOutputs = 1; mbqNew = minibatchqueue(dsTest,numOutputs,... MiniBatchSize=miniBatchSize, ... MiniBatchFcn=@preprocessMiniBatchPredictors, ... MiniBatchFormat="CBT", ... OutputEnvironment=executionEnvironment);
0과 5 사이의 균일한 간격의 타임스탬프 1000개를 목표 타임스탬프로 지정합니다.
tspanTarget = linspace(0,5,1000);
루프를 사용해 미니 배치 대기열을 순회하여 예측을 수행합니다.
YNew = modelPredictions(parameters,tspan,mbqNew,tspanTarget);
예측값으로 구성된 배열 크기를 확인합니다.
size(YNew)
ans = 1×3
1 200 1000
산점도 플롯으로 입력값을 플로팅한 다음 예측된 시퀀스를 플로팅하여 첫 번째 예측값을 시각화합니다.
입력 데이터를 플로팅합니다.
idx = 1; X = sequencesTest(:,idx,:); figure t = tiledlayout(numChannels,1); title(t,"Input Sequence") for i = 1:numChannels nexttile scatter(tspan,squeeze(X)) xlabel("t") ylabel("Value") end
예측된 값을 플로팅합니다.
for i = 1:numChannels nexttile(i) hold on plot(tspanTarget,squeeze(YNew(i,idx,:))); end title(t,"Predicted Sequence") legend(["Input Data" "Prediction"],Location="southeast");

지원 함수
모델 함수
model 함수(이 예제의 모델 함수 정의하기 섹션에 소개되어 있음)는 모델의 학습 가능한 파라미터, 소스 타임스탬프 tspan 및 그에 대응되는 시퀀스 값과 마스크, 목표 타임스탬프 tspanTarget을 입력값으로 받습니다. 이 함수는 목표 타임스탬프에 대응되는 예측값 Y를 반환합니다.
다음 도식은 모델 함수의 구조를 보여줍니다.
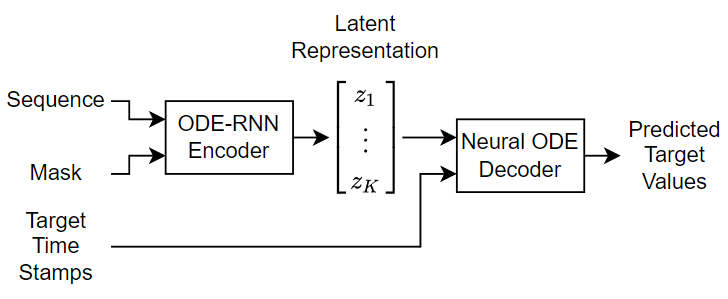
신경망 ODE 디코더는 잠재 표현으로 인코딩된 평균과 분산 값을 갖는 가우스 분포에서 샘플링하여 입력 시퀀스를 복원합니다.
function Y = model(parameters,tspan,X,tspanTarget,args) arguments parameters tspan X tspanTarget args.Mask = dlarray(true(size(X)),"CBT") end mask = args.Mask; Z = encoder(parameters.Encoder,tspan,X,Mask=mask); % Split the latent representation into mean and variance. latentSize = size(Z,1)/2; mu = Z(1:latentSize,:); sigma = abs(Z(latentSize+(1:latentSize),:)); % Take samples of the latent distribution. epsilon = randn(size(mu),like=X); Z = epsilon.*sigma + mu; Z = dlarray(Z,"CB"); % Decode the latent representation. Y = decoder(parameters.Decoder,tspanTarget,Z); end
인코더 함수
encoder 함수(이 예제의 모델 함수 정의하기 섹션에 소개되어 있음)는 인코더의 학습 가능한 파라미터, 소스 타임스탬프 tspan 및 그에 대응되는 시퀀스 값과 마스크를 입력값으로 받습니다. 이 함수는 잠재 표현을 출력합니다.
다음 도식은 인코더의 구조를 보여줍니다.
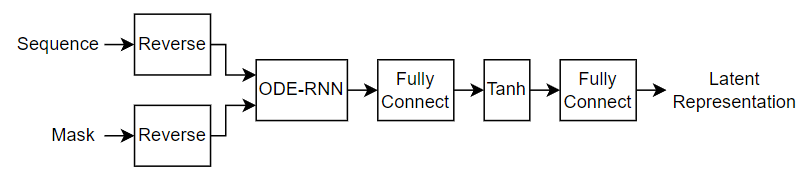
이 인코더는 인코더에 대한 마지막 입력값이 디코더에 대한 첫 번째 출력값이 되도록 입력 시퀀스의 순서를 뒤집습니다. ODE-RNN은 순서를 뒤집은 데이터의 각 시간 스텝을 마스크 처리된 게이트 순환 유닛(GRU)과 ODE 솔버를 사용해 읽어 들이면서 잠재 표현을 업데이트합니다. ODE-RNN은 입력 데이터와 GRU 연산의 은닉 상태를 결합하고, 신경망 ODE(Neural ODE)를 기반으로 한 GRU 연산의 출력값을 다음 시간 스텝으로 진전시킵니다. GRU 연산은 마스크로 지정된 시간 스텝에서만 업데이트된 상태를 사용합니다.
다음 도식은 데이터의 시간 스텝을 처리할 때의 신경망 ODE-RNN 구조를 보여줍니다. 이 도식에서 는 시간 스텝의 값을 나타내고, 는 마스크 값을 나타내며, 는 GRU 연산의 은닉 상태 출력값입니다. ODE 솔버 스텝은 성능을 위한 간단한 고정 스텝 오일러 방법입니다.
function Z = encoder(parameters,tspan,X,args) arguments parameters tspan X args.Mask = dlarray(true(size(X)),"CBT") end mask = args.Mask; % Reverse time. tspan = flip(tspan,2); X = flip(X,3); mask = flip(mask,3); % Initialize the hidden state for the RNN. hiddenSize = size(parameters.gru.RecurrentWeights,2); [~,batchSize, sequenceLength] = size(X); h = zeros(hiddenSize,batchSize,like=X); h = dlarray(h,"CB"); latentSize = size(parameters.ODE.fc1.Weights,2); % Solve the ODE-RNN in a loop. for t = 1:sequenceLength-1 ZPrev = h(1:latentSize,:); % Solve the ODE. Zt = euler(@odeModel,[tspan(t) tspan(t+1)],ZPrev,parameters.ODE); % Concatenate the input data with the RNN input over the channel dimension. Zt = dlarray(Zt,"CBT"); Xt = X(:,:,t); Zt = cat(1,Zt,Xt); % RNN step. inputWeights = parameters.gru.InputWeights; recurrentWeights = parameters.gru.RecurrentWeights; bias = parameters.gru.Bias; [Z,hnew] = gru(Zt,h,inputWeights,recurrentWeights,bias); % Update the RNN state where the data is not missing. h = hnew.*mask(:,:,t) + h.*(1-mask(:,:,t)); end % Apply output transformation. weights = parameters.fc1.Weights; bias = parameters.fc1.Bias; Z = fullyconnect(Z,weights,bias); Z = tanh(Z); weights = parameters.fc2.Weights; bias = parameters.fc2.Bias; Z = fullyconnect(Z,weights,bias); end
디코더 함수
decoder 함수(이 예제의 모델 함수 정의하기 섹션에 소개되어 있음)는 디코더의 학습 가능한 파라미터, 목표 타임스탬프 tspanTarget 및 잠재 표현 Z를 입력값으로 받습니다. 이 함수는 목표 타임스탬프에 대응되는 예측값을 반환합니다.
다음 도식은 디코더의 구조를 보여줍니다.

function Y = decoder(parameters,tspanTarget,Z) % Apply the neural ODE operation. Y = dlode45(@odeModel,tspanTarget,Z,parameters.ODE, ... RelativeTolerance=1e-3, ... AbsoluteTolerance=1e-4); % Concatenate over the time dimension. Z = dlarray(Z,"CBT"); Y = cat(3,Z,Y); % Apply the fully connect operation. weights = parameters.fc.Weights; bias = parameters.fc.Bias; Y = fullyconnect(Y,weights,bias); end
모델 손실 함수
modelLoss 함수는 모델의 학습 가능한 파라미터, 소스 타임스탬프 tspan 및 그에 대응되는 시퀀스 값과 마스크를 입력값으로 받습니다. 이 함수는 모델 손실과 학습 가능한 파라미터에 대한 손실 기울기를 반환합니다.
모델 손실 함수는 입력 관측값 수과 샘플 수에 대해 정규화된 손실을 사용합니다.
function [loss,gradients] = modelLoss(parameters,tspan,X,mask,T) % Model forward pass. tspanDecoder = tspan; Y = model(parameters,tspan,X,tspanDecoder,Mask=mask); % Reconstruction loss. loss = l2loss(Y,T,Reduction="none"); % Normalize by the number of non-missing elements. loss = sum(loss,[1 3]) ./ sum(mask,[1 3]); loss = mean(loss); % Gradients. gradients = dlgradient(loss,parameters); end
모델 예측 함수
modelPredictions 함수는 모델의 학습 가능한 파라미터, 소스 타임스탬프 tspan, 데이터로 구성된 미니 배치 대기열 및 목표 타임스탬프 tspanTarget을 입력값으로 받습니다. 이 함수는 모델 예측값 Y를 반환합니다.
function Y = modelPredictions(parameters,tspan,mbq,tspanTarget) Y = []; while hasdata(mbq) % Read mini-batch of validation data. X = next(mbq); % Model forward pass. YBatch = model(parameters,tspan,X,tspanTarget); Y = cat(2,Y,YBatch); end end
ODE 모델 함수
함수 odeModel은 함수 입력값 t(사용되지 않음)와 y, 그리고 컨벌루션 가중치와 편향을 포함하는 ODE 함수 파라미터를 입력값으로 받습니다. 이 함수는 3개의 완전 연결 계층 간에 tanh 연산이 있는 신경망의 출력값을 반환합니다.
인코더와 디코더는 신경망 ODE를 사용합니다. 신경망 ODE는 형식의 ODE 문제이고, 여기서 는 입력값 와 학습 가능한 파라미터 를 갖는 신경망입니다. 이 경우 인코더와 디코더의 신경망 ODE는 동일한 신경망 를 사용하는데, 이 신경망은 tanh 활성화가 사이에 포함된 3개의 완전 연결 연산으로 구성됩니다.
다음 도식은 신경망의 구조를 보여줍니다.

function z = odeModel(~,y,parameters) weights = parameters.fc1.Weights; bias = parameters.fc1.Bias; z = fullyconnect(y,weights,bias); z = tanh(z); weights = parameters.fc2.Weights; bias = parameters.fc2.Bias; z = fullyconnect(z,weights,bias); z = tanh(z); weights = parameters.fc3.Weights; bias = parameters.fc3.Bias; z = fullyconnect(z,weights,bias); end
미니 배치 전처리 함수
preprocessMiniBatch 함수는 다음 단계를 사용하여 데이터를 전처리합니다.
preprocessMiniBatchPredictors함수를 사용하여 예측 변수를 전처리합니다.입력 데이터와 일치하는, 목표값으로 구성된 미니 배치를 만듭니다.
시퀀스 데이터로 구성된 시간 스텝 50개를 무작위로 0으로 설정하고, 누락값을 나타내는 마스크를 만듭니다.
function [X,mask,T] = preprocessMiniBatch(XCell) X = preprocessMiniBatchPredictors(XCell); mask = true(size(X)); T = X; % Remove time steps at random. [~,numObservations,numTimestamps] = size(X); for n = 1:numObservations idx = randsample(numTimestamps,50); idx = sort(idx); X(:,n,idx) = 0; mask(:,n,idx) = false; end end
미니 배치 예측 변수 전처리 함수
preprocessMiniBatchPredictors 함수는 입력 셀형 배열에서 시퀀스 데이터를 추출하여 미니 배치 예측 변수를 전처리하고, 두 번째 차원을 따라 내용을 결합하여 숫자형 배열로 변환합니다.
function X = preprocessMiniBatchPredictors(XCell) X = cat(2,XCell{:}); end
순방향 오일러 솔버
euler 함수는 ODE 함수 f, 시간 구간 t, 입력값 y, ODE 파라미터, 그리고 구간을 반복할 때의 스텝 크기를 지정하는 선택적 이름-값 인수 MaxStepSize를 입력값으로 받습니다. 이 함수는 순방향 오일러 출력값을 반환합니다. 순방향 오일러 함수는 속도가 빠른 ODE 솔버이지만, dlode45 같은 적응형 ODE 솔버보다 정확도와 유연성이 대체로 떨어집니다.
function y = euler(f,t,y,parameters,args) arguments f t y parameters args.StepSize = 0.1; end stepSize = args.StepSize; t1 = t(1); t2 = t(2); t2 = min(t2,t1 - stepSize); tspan = t1:stepSize:t2; y = y; for i = 1:numel(tspan)-1 y = y + (t(i+1)-t(i))*f(t,y,parameters); end end
참고 문헌
[1] Chen, Ricky T. Q., Yulia Rubanova, Jesse Bettencourt, and David Duvenaud. “Neural Ordinary Differential Equations.” Preprint, submitted December 13, 2019. https://arxiv.org/abs/1806.07366
[2] Yulia Rubanova, Ricky T. Q. Chen, David Duvenaud. "Latent ODEs for Irregularly-Sampled Time Series" Preprint, submitted July 8, 2019. https://arxiv.org/abs/1907.03907
[3] Diederik P Kingma, Max Welling. "Auto-Encoding Variational Bayes." Preprint, submitted, submitted December 20, 2013. https://arxiv.org/abs/1312.6114
참고 항목
dlode45 | dlarray | dlfeval | dlgradient | fullyconnect | minibatchqueue | l2loss | gru